Nat. Commun. | 利用受限玻尔兹曼机设计分子RNA开关
Nat. Commun. | 利用受限玻尔兹曼机设计分子RNA开关

DrugOne
发布于 2025-12-25 15:05:36
发布于 2025-12-25 15:05:36
DRUGONE
核糖开关是一类能够在配体结合后发生构象变化、从而调控基因表达的结构化 RNA 分子。研究人员提出一种基于受限玻尔兹曼机(Restricted Boltzmann Machine, RBM)的生成式方法,从天然同源序列中学习序列–结构–功能约束,用于从零开始设计具有构象切换能力的分子 RNA 开关。以 SAM-I 核糖开关适配体为例,RBM 不仅能够重现天然序列的保守性、协变关系和多样性,还能生成与天然序列差异显著的新序列。对近 500 条设计序列的高通量化学探针实验表明,高评分 RBM 设计序列中约 30–35% 能在 SAM 存在下发生与天然核糖开关相似的结构切换,揭示了 RNA 构象切换与局部能量窗口之间的内在联系。

RNA 的序列到结构再到功能的映射高度复杂,尤其是核糖开关这类依赖多重稳定构象与配体诱导切换的 RNA 元件。传统理性设计方法多基于最低自由能二级结构,往往难以处理假结和三维相互作用;定向进化方法虽然有效,但实验成本高、探索空间受限。随着大规模同源 RNA 序列数据库的积累,如何利用进化信息构建能够生成具有真实功能的 RNA 序列模型,成为 RNA 设计领域的核心挑战。
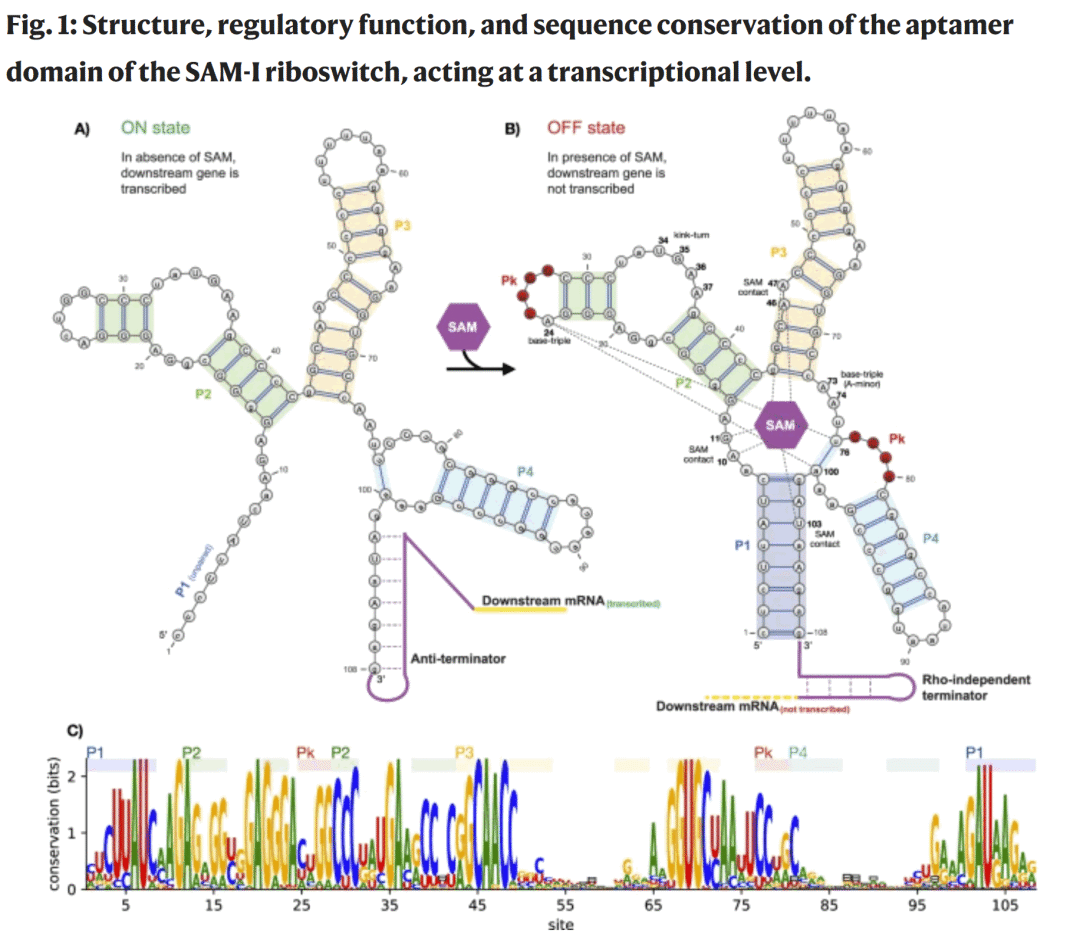
图1| SAM-I 核糖开关适配体的结构、功能与序列保守性。
方法
研究人员在 SAM-I 核糖开关适配体的多序列比对数据上训练 RBM,将 RNA 序列建模为显层变量,通过隐层学习跨位点的高阶统计依赖。RBM 由此隐式编码了二级结构、假结及关键三级相互作用的约束。模型生成的新序列通过 SHAPE 和 DMS 两种化学探针方法进行高通量实验验证,并结合统计分析框架,量化配体诱导的整体构象变化能力。
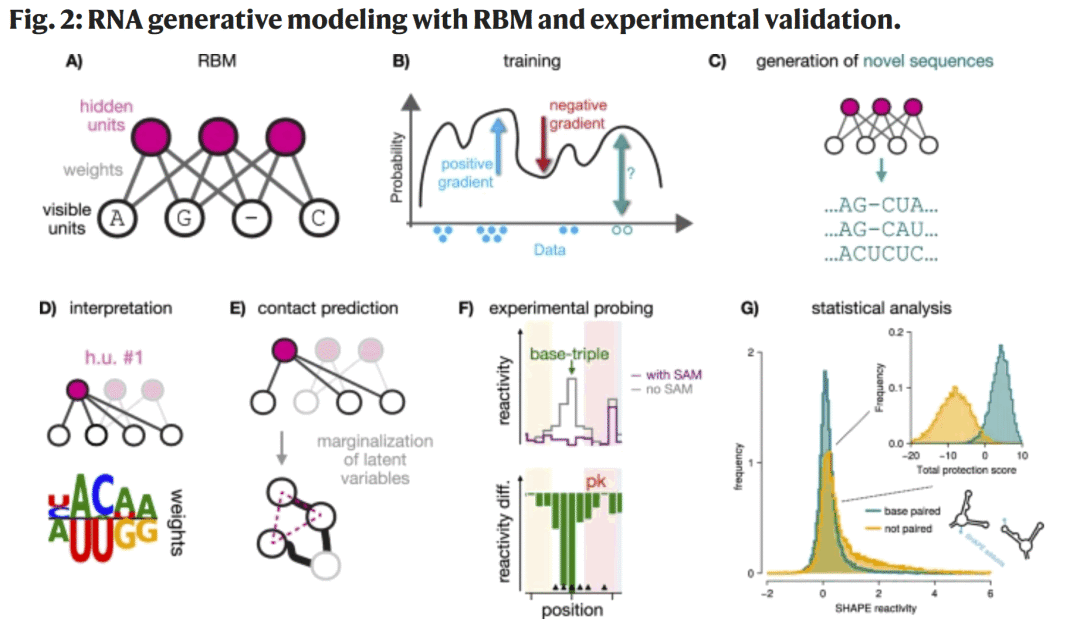
图2| 基于 RBM 的 RNA 生成与实验验证流程。
结果
RBM 学习并重现天然核糖开关的结构约束
RBM 从序列统计中自动识别出与螺旋、假结和三级接触相关的关键特征,其推断的位点相互作用与已知结构高度一致,证明模型能够捕获超越简单二级结构的高阶约束。
RBM 生成序列覆盖天然多样性空间
与协方差模型相比,RBM 生成序列在主成分空间中更好地覆盖天然序列的多样性,能够产生远离已知序列、但仍满足功能约束的候选 RNA。
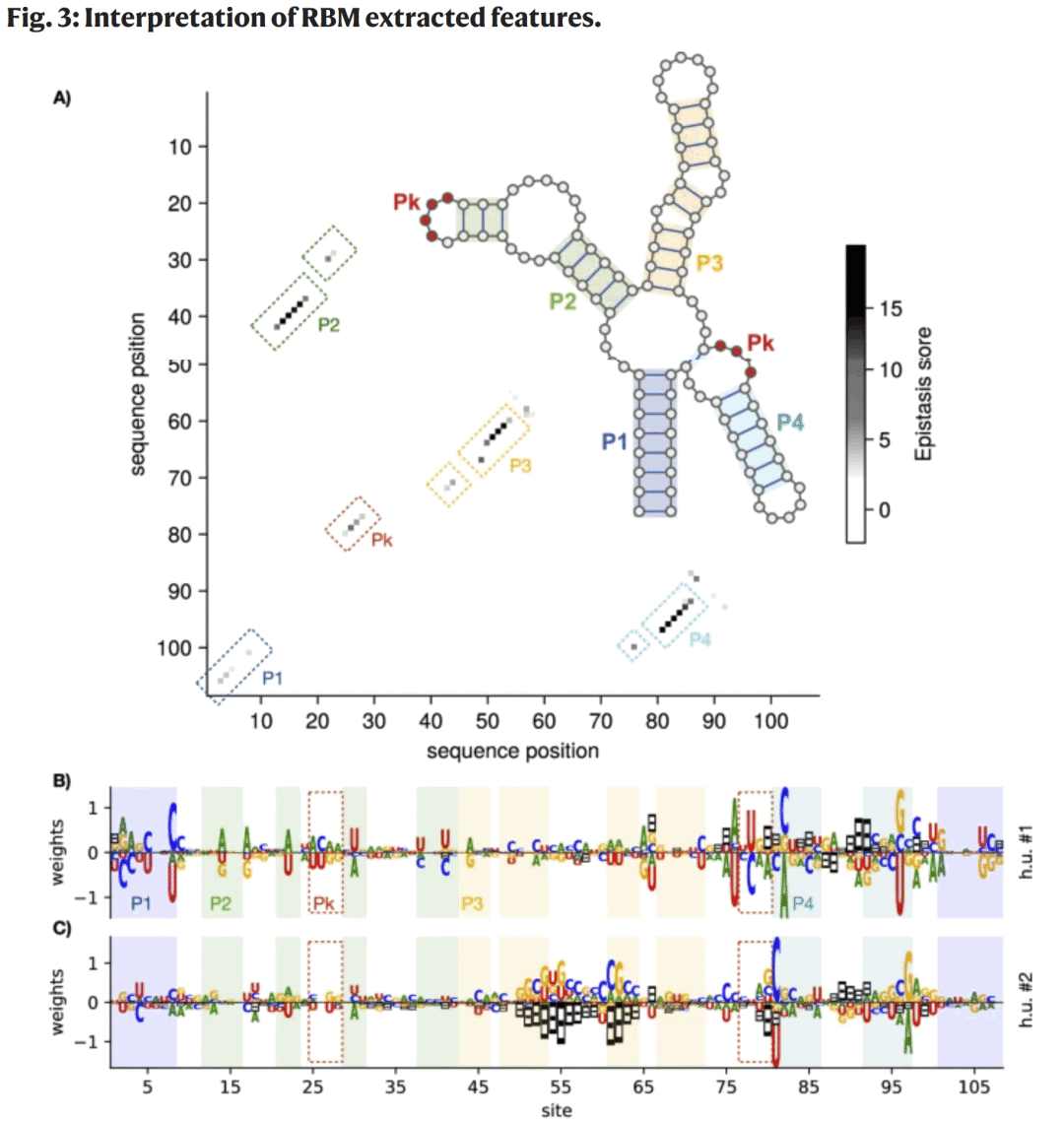
图3| RBM 学习到的结构与序列特征解释。
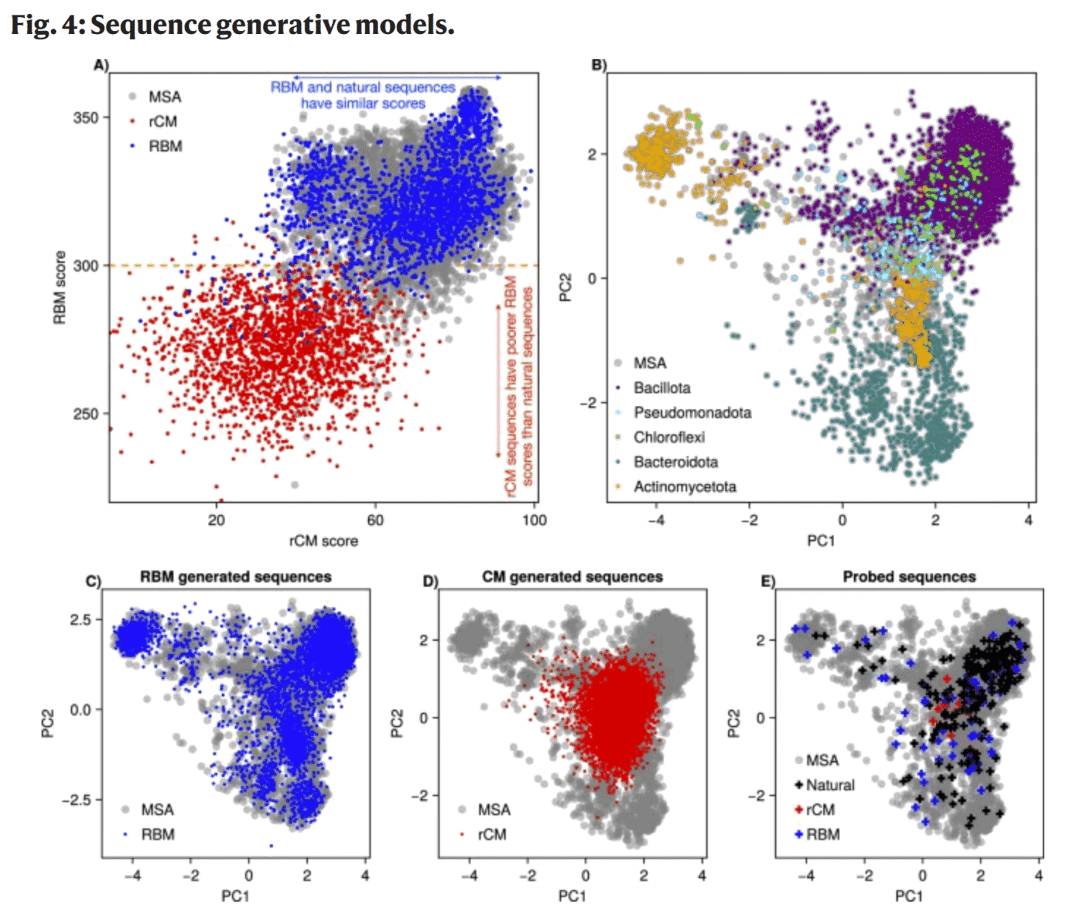
图4| RBM 与协方差模型生成序列的多样性对比。
设计序列的结构切换实验验证
在 476 条 RBM 设计序列中,高评分序列表现出与天然适配体相似的 SAM 诱导构象变化,其化学反应性变化集中于配体结合位点、假结和关键螺旋区域。
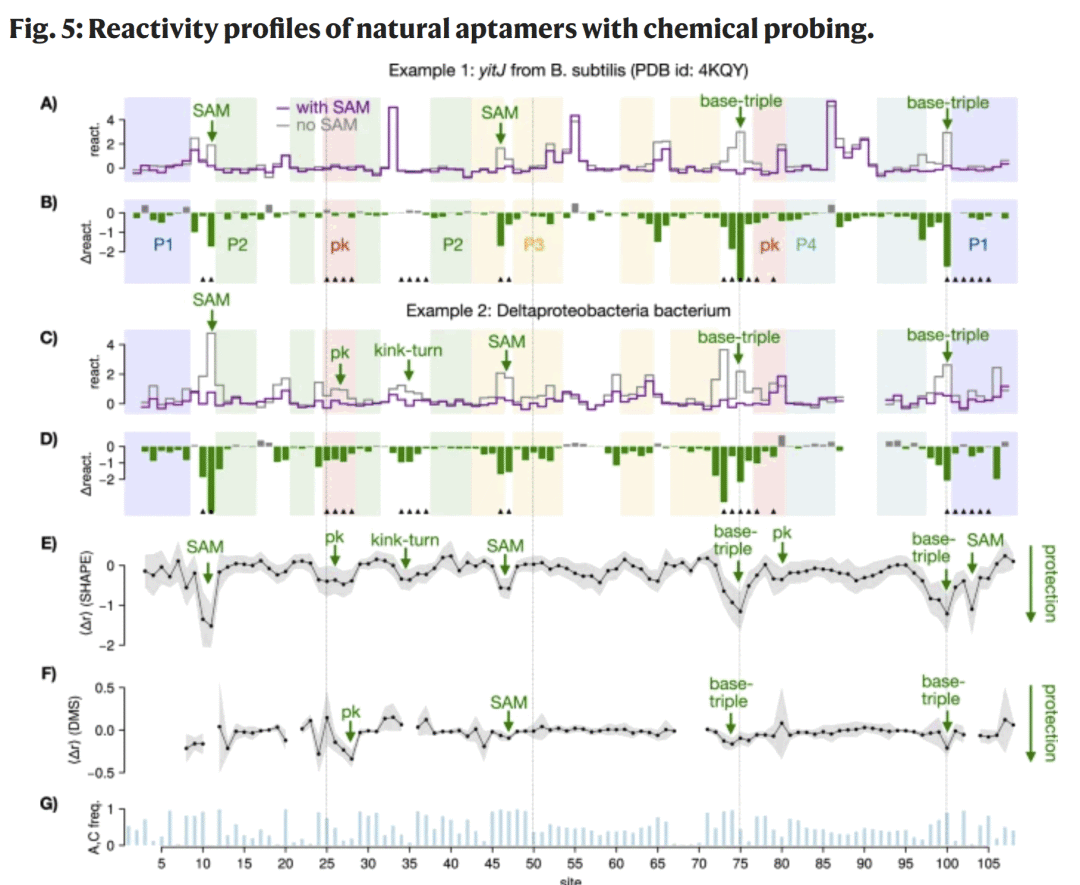
图5| 天然适配体的化学探针反应性特征。
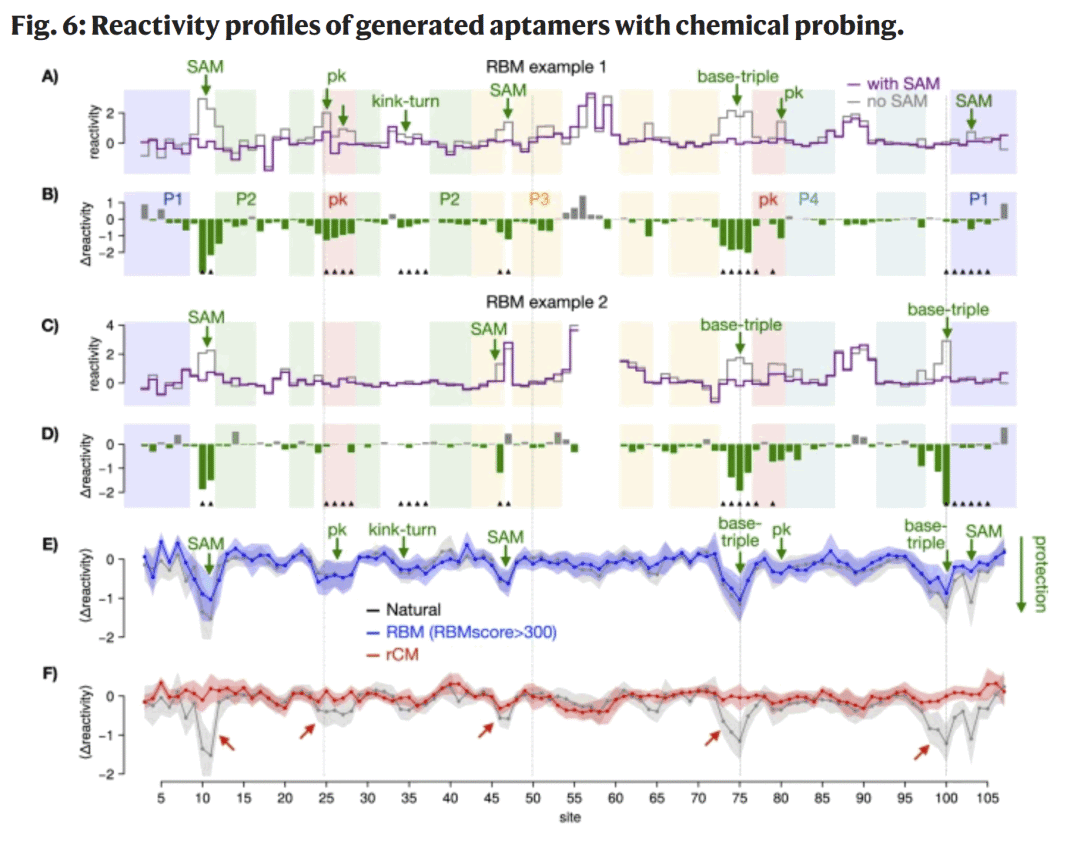
图6| RBM 设计适配体的结构响应对比。
统计框架定量评估 RNA 开关功能
通过定义聚合多位点信息的“保护评分”,研究人员将 RNA 分为响应型、非响应型和不确定型。RBM 高评分序列显著富集于响应型,而协方差模型生成序列几乎不表现出有效切换。
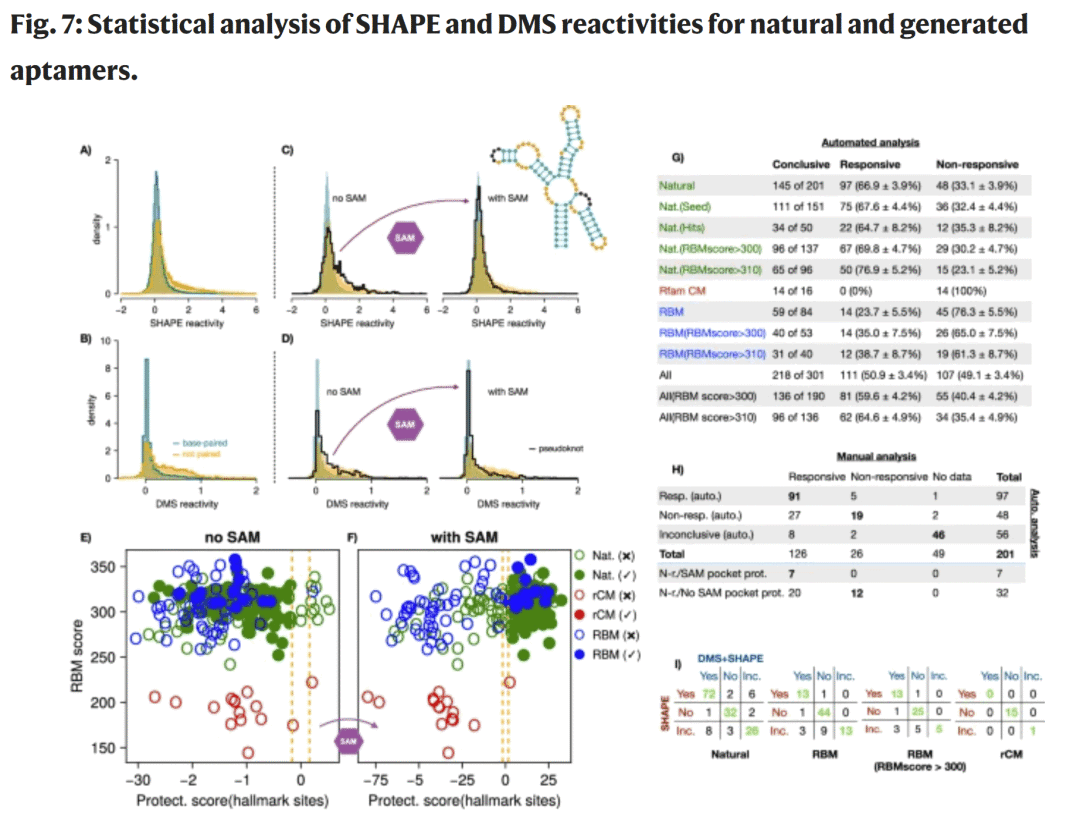
图7| 天然与设计序列的统计判别分析。
构象切换依赖于中等稳定性的能量窗口
分析显示,能够发生切换的 RNA 在关键螺旋和假结区域具有中等配对能量,既避免过度稳定,也防止结构过于松散。RBM 生成序列天然偏向这一能量窗口,从而提高了功能成功率。
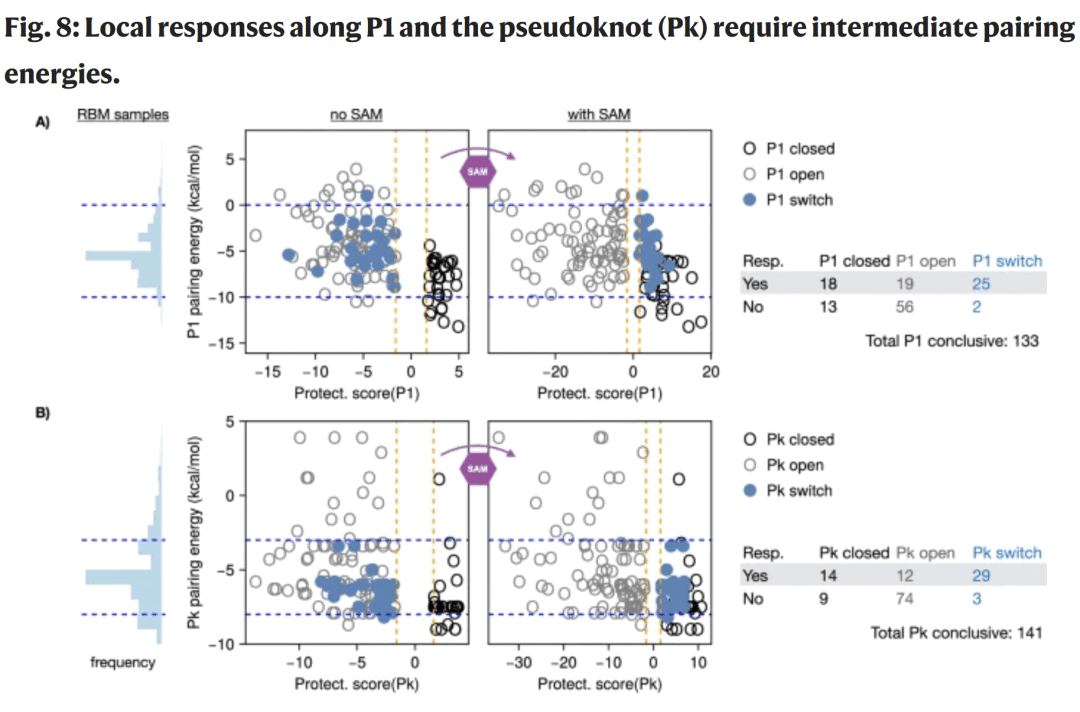
图8| 局部能量窗口与构象切换能力的关系。
扩展设计验证与模型泛化能力
在进一步的设计–实验循环中,RBM 仍能生成高比例可切换 RNA,包括缺失某些螺旋或与天然序列差异极大的新序列,显示出良好的泛化能力。
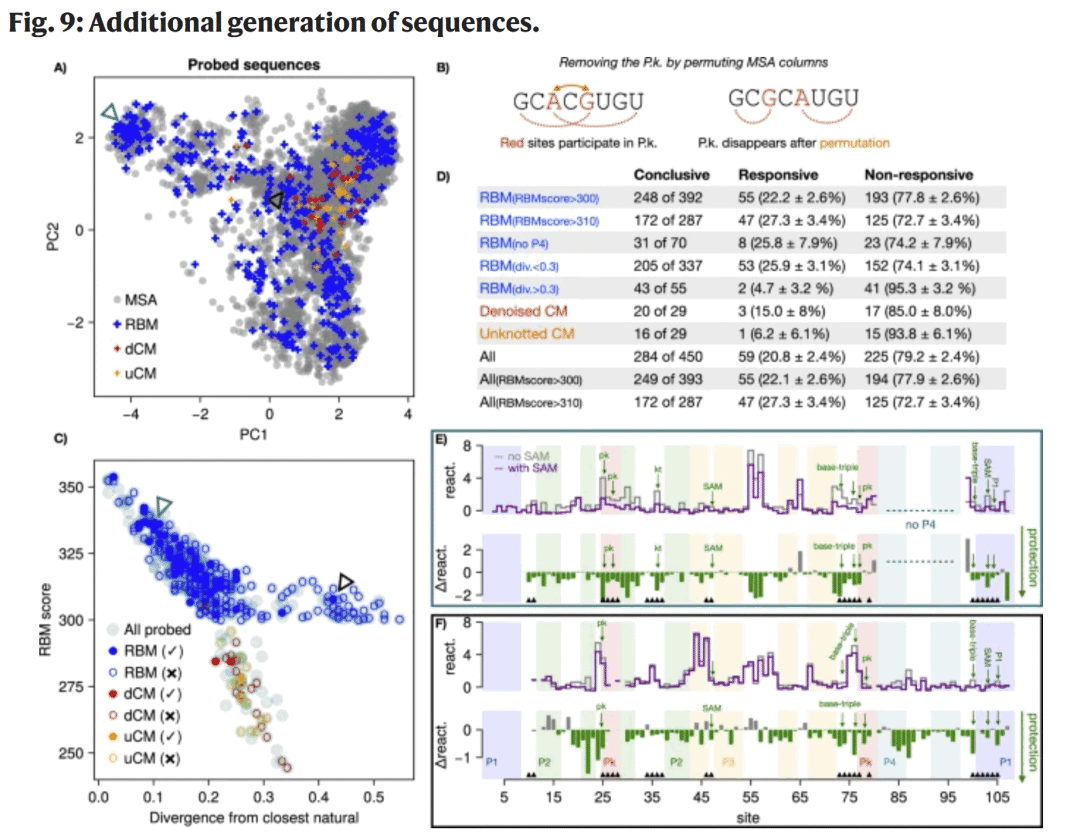
图9| 扩展设计实验与远离天然序列的功能性 RNA 。
讨论
该研究表明,RBM 能够作为一种可解释、有效的生成模型,直接从进化序列数据中学习 RNA 构象切换所需的复杂约束,并用于功能性 RNA 开关的设计。与传统基于二级结构的理性设计或协方差模型相比,RBM 在捕获假结和三级相互作用方面具有显著优势。尽管当前成功率仍低于天然序列水平,但该方法首次证明了无需显式结构建模即可设计具备变构功能的 RNA 分子,为 RNA 合成生物学、分子传感器及可编程调控元件的设计提供了新路径。
整理 | DrugOne团队
参考资料
Fernandez-de-Cossio-Diaz, J., Hardouin, P., Lyonnet du Moutier, FX. et al. Designing molecular RNA switches with Restricted Boltzmann machines. Nat Commun 16, 11223 (2025).
https://doi.org/10.1038/s41467-025-66265-y
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-23,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

