IGV查看拷贝数变异需要的segment文件格式解析
IGV查看拷贝数变异需要的segment文件格式解析
有小伙伴委托我们做肿瘤外显子数据分析,主要是后面的统计可视化部分,因为前面的测序fastq文件基本上公司就随便走流程拿到了snp或者indel,如果是肿瘤外显子通常是maf格式的somatic突变信息文件。
但是绝大部分小伙伴其实并不会给很标准的文件格式给我们,还好我们的数据清洗技术还不错,就是费一点功夫,慢慢整理每个样品的snp或者indel,基本过滤和格式转换后,就可以出全景图,比如2021的文章:《Correlation of mutational landscape and survival outcome of peripheral T-cell lymphomas》,就是
- 测序:150-bp pair-end reads on the NovaSeq
- 比对:Sequence reads were aligned using BWA
- 找突变SNVs :Single nucleo- tide variants (SNVs) were called using MuTect
- 找突变Indels:Small insertions and deletions (Indels) were determined by GATK
有了Indels和SNV就可以进行如下所示的肿瘤队列突变全景图:
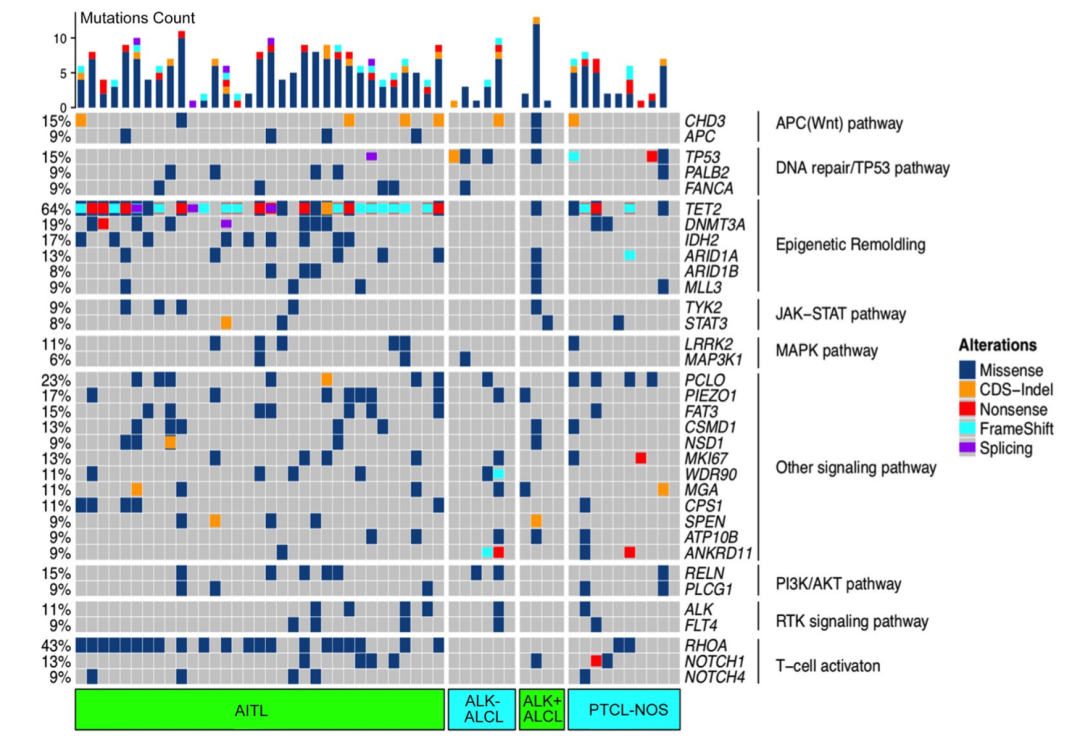
肿瘤队列突变全景图
但是它仅仅是Indels和SNV,并不是拷贝数变异信息,IGV查看拷贝数变异需要的segment文件格式。巧妇难为无米之炊,如果不给我们segment文件格式拷贝数变异信息记录文件,我们没办法进行可视化的。比如文章:《Patient-Derived Organoids Can Guide Personalized-Therapies for Patients with Advanced Breast Cancer》,就是挑选了几个病人进行类似于IGV一样的CNV可视化:

类似于IGV一样的CNV可视化
那它到底是需要什么样的文件呢?其实IGV软件就给出来了标准,而且有一个示例文件:
'ID chrom loc.start loc.end num.mark seg.mean
GenomeWideSNP_416532 1 51598 76187 14 -0.7116
GenomeWideSNP_416532 1 76204 16022502 8510 -0.029
GenomeWideSNP_416532 1 16026084 16026512 6 -2.0424
GenomeWideSNP_416532 1 16026788 17063449 424 -0.1024
GenomeWideSNP_416532 1 17067742 17134834 61 -0.6868
GenomeWideSNP_416532 1 17148828 17965202 543 0.0072
GenomeWideSNP_416532 1 17971140 17977142 2 -2.3959
GenomeWideSNP_416532 1 17977404 25455928 4786 -0.0199
就是标准的6列:
- 第一列是样品名字,每个segment文件可以有多个样品。
- 第二列是染色体,一般来说就是 1-22号染色体,性染色体通常是不需要看的
- 第三列和第四列就是每个 拷贝数变异区域的起始终止坐标,拷贝数变异范围动态变化很大,几百万个碱基是很正常的。
- 第五列是可有可无的,至少IGV软件是可以忽略它,后续的gistic软件也似乎是不需要这个信息
- 第六列是重点,拷贝数变化的情况,0代表是正常的二倍体,一般来说正负0.3范围内都是可以接受的接近二倍体。
有了这样的segment文件格式拷贝数变异信息记录文件,基本上你看到的文献里面的图表,我们都可以帮忙制作 出来。
参考:
- 官方文档1 https://software.broadinstitute.org/software/igv/SegmentedData
- 官方文档2 https://software.broadinstitute.org/software/igv/SEG
- IGV效果:https://software.broadinstitute.org/software/igv/DefaultDisplay
- 文件案例:https://software.broadinstitute.org/software/igv/sites/cancerinformatics.org.igv/files/linked_files/example.seg
腾讯云开发者

扫码关注腾讯云开发者
领取腾讯云代金券
Copyright © 2013 - 2025 Tencent Cloud. All Rights Reserved. 腾讯云 版权所有
深圳市腾讯计算机系统有限公司 ICP备案/许可证号:粤B2-20090059 深公网安备号 44030502008569
腾讯云计算(北京)有限责任公司 京ICP证150476号 | 京ICP备11018762号 | 京公网安备号11010802020287
Copyright © 2013 - 2025 Tencent Cloud.
All Rights Reserved. 腾讯云 版权所有