MYSQL 性能优化 index 函数,隐藏,前缀,hash 索引 使用方法(2)
MYSQL 性能优化 index 函数,隐藏,前缀,hash 索引 使用方法(2)

接着上期说,MYSQL 8 的索引的方式相对MYSQL 5.7 有了很多的进步,其中最突出的就是添加的functional indexes , 那么这个功能到底是什么时候开始的有的。
8.013 ,对MYSQL 8.013版本中开始添加了函数索引,下面我们举一个例子
create table function_test (id bigint primary key auto_increment, name varchar(20) not null, birth_day datetime);

mysql> insert into function_test (name,birrth_day) values ('Tim','2018-09-09'); Query OK, 1 row affected (0.01 sec) mysql> insert into function_test (name,birrth_day) values ('Sam','2020-01-09'); Query OK, 1 row affected (0.01 sec) mysql> insert into function_test (name,birrth_day) values ('Pam','2021-01-09'); Query OK, 1 row affected (0.00 sec) Alter table function_test add index month(birrth_day);

实际上我们得到了一个带有函数的索引 CREATE TABLE `function_test` ( `id` bigint NOT NULL AUTO_INCREMENT, `name` varchar(20) NOT NULL, `birrth_day` datetime DEFAULT NULL, PRIMARY KEY (`id`), KEY `month` (`birrth_day`) ) ENGINE=InnoDB AUTO_INCREMENT=4 DEFAULT CHARSET=utf8mb4 COLLATE=utf8mb4_0900_ai_ci |
explain select birrth_day from function_test where month(birrth_day) = 5; explain select birrth_day from function_test where month(birrth_day) = 5;

基于这个功能,MYSQL的的版本应该是在8.013 版本以上。
而前缀索引是MYSQL独有的功能,但使用时有场景的,也就是首先添加前缀索引的额类型是字符类型,通过字段前部分的字段的值就可以明细查找数据,其主要的目的就是减少索引字节的同时,还能提高效率。
我们举例
create table prefix_test (id bigint primary key auto_increment,name varchar(20) not null, type_comments varchar(2000));


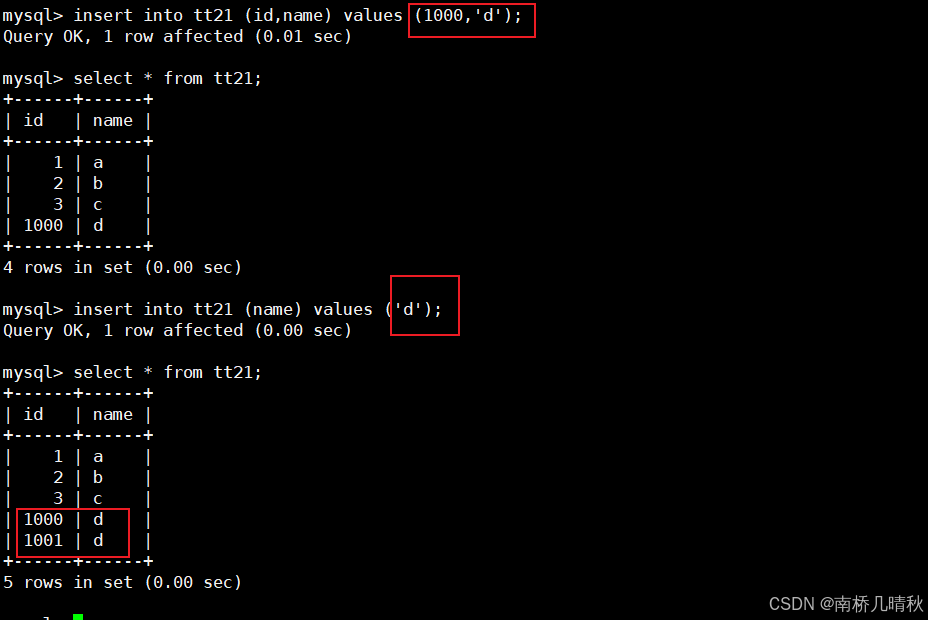
mysql> insert into prefix_test (name,type_comments) values ('Tim','1_我不喜欢'); Query OK, 1 row affected (0.01 sec) mysql> insert into prefix_test (name,type_comments) values ('Sam','3_我很喜欢'); Query OK, 1 row affected (0.01 sec) mysql> insert into prefix_test (name,type_comments) values ('Semon','2_没感觉'); Query OK, 1 row affected (0.00 sec) mysql> alter table prefix_test add index (type_comments(1)); Query OK, 0 rows affected (0.07 sec) Records: 0 Duplicates: 0 Warnings: 0
explain select name from prefix_test where type_comments like '1%'; 通过上面的例子也可以看出,实际上我们只需要给2000 字符的,中的一个1个字符建立索引,就可以完成某些快速查找的完成。

或者可以通过这样的方式进行GROUP BY 等方式的数据组织等。
在这些过后就是MYSQL 的隐藏索引,这个并不是一个新的功能,在其他数据库也有类似的索引类型,实际上隐藏索引是针对某些查询来验证索引的有效性,并且让他上线或者下线的功能。

mysql> mysql> alter table prefix_test add index (type_comments(1)) invisible; Query OK, 0 rows affected (0.08 sec) Records: 0 Duplicates: 0 Warnings: 0 mysql> show create table prefix_test; CREATE TABLE `prefix_test` ( `id` bigint NOT NULL AUTO_INCREMENT, `name` varchar(20) NOT NULL, `type_comments` varchar(2000) DEFAULT NULL, PRIMARY KEY (`id`), KEY `type_comments` (`type_comments`(1)) /*!80000 INVISIBLE */ ) ENGINE=InnoDB AUTO_INCREMENT=4 DEFAULT CHARSET=utf8mb4 COLLATE=utf8mb4_0900_ai_ci | mysql> explain select name from prefix_test where type_comments like '1%'; | id | select_type | table | partitions | type | possible_keys | key | key_len | ref | rows | filtered | Extra | | 1 | SIMPLE | prefix_test | NULL | ALL | NULL | NULL | NULL | NULL | 3 | 33.33 | Using where | 1 row in set, 1 warning (0.00 sec) 上面的图中可以展示出虽然建立了新的索引但是invisible 的状态是无法使用的。如果要是要使用可以立即让他进入到 visible的状态。
最后是hash 索引,在上一篇中提到了自己建立了HASH 的方式解决等值索引中的多个字段建立联合索引,我们下面举一个例子来看看如何操作。
mysql> alter table hash_test add hash_col varchar(20) generated always as (crc32(concat(type1,type2))); Query OK, 0 rows affected (0.03 sec) Records: 0 Duplicates: 0 Warnings: 0 mysql> select * from hash_test; +----+------+-------+-------+------------+ | id | name | type1 | type2 | hash_col | +----+------+-------+-------+------------+ | 1 | Tim | 10 | 20 | 2249310613 | +----+------+-------+-------+------------+ 1 row in set (0.00 sec) mysql> insert into hash_test (name,type1,type2) values ('Sam','20','30'); Query OK, 1 row affected (0.00 sec) mysql> select * from hash_test; +----+------+-------+-------+------------+ | id | name | type1 | type2 | hash_col | +----+------+-------+-------+------------+ | 1 | Tim | 10 | 20 | 2249310613 | | 2 | Sam | 20 | 30 | 2378129210 | +----+------+-------+-------+------------+ 2 rows in set (0.00 sec)

下面我们就可以给hash 的虚拟字段建立索引

那么下次我们下次在查询的时候,就可以直接使用hash_col 来作为查找的索引字段,而不再关心ytype1 type2 如果有50个字段要建立索引,那么也可以不管,就一个字段可以表达N 个字段的值,这样查询速度快,同时你的索引任何时刻只有一个,空间效率的问题都解决了。
本文分享自 AustinDatabases 微信公众号,前往查看
如有侵权,请联系 cloudcommunity@tencent.com 删除。
本文参与 腾讯云自媒体同步曝光计划 ,欢迎热爱写作的你一起参与!
腾讯云开发者

扫码关注腾讯云开发者
领取腾讯云代金券
Copyright © 2013 - 2025 Tencent Cloud. All Rights Reserved. 腾讯云 版权所有
深圳市腾讯计算机系统有限公司 ICP备案/许可证号:粤B2-20090059 深公网安备号 44030502008569
腾讯云计算(北京)有限责任公司 京ICP证150476号 | 京ICP备11018762号 | 京公网安备号11010802020287
Copyright © 2013 - 2025 Tencent Cloud.
All Rights Reserved. 腾讯云 版权所有