NLS曲线拟合奇异矩阵误差
NLS曲线拟合奇异矩阵误差
提问于 2017-02-28 06:24:43
我需要帮助拟合nls和找到初步估计,不会导致奇异矩阵。我将非常感谢任何帮助。
via_data$Concentration <- c(0.197, 0.398, 0.792, 1.575,
3.154, 6.270, 12.625, 25.277,
25.110, 49.945, 74.680)
via_data$Viability <- c(100, 94.62, 96.21, 87.53,
80, 62.22, 39.11,
30.80, 30, 22, 2.56)
x <- via_data$Concentration
y <- via_data$Viability
fit <- nls(y ~((1/(1+Epsup/x)^Bup)*(1/(1+Epsdn/x)^Bdn)), start=list(Epsup=0, Bup=1, Epsdn=10, Bdn=-5), trace=T)
Error in nlsModel(formula, mf, start, wts) :
singular gradient matrix at initial parameter estimates谢谢你,克里娜
回答 1
Stack Overflow用户
回答已采纳
发布于 2017-02-28 07:44:32
在st下面是您的起始值,但我们使用Epsup=1来避免0的简并。fo是公式。为了防止将负数提高到幂,我们用sqrt(Epsup^2)代替了sqrt(Epsup^2),对于Epsdn也是如此--这增加了Epsup和Espdn不能是负的假设。(这与使用abs(Epsup)相同;但是,nlxb的派生表中没有abs。)接下来,使用nls2在st/10和10*st边界之间的网格上生成值。nls2将生成这些对象,并返回一个"nls"对象,并找到最好的对象。现在使用它作为nlmrt包的nlxb的起始值。它比nls更好地处理困难的问题。nlxb不返回"nls"对象(虽然包中有运行nlxb的wrapnls,然后是nls,但是我们不能从nlxb获得直接输出),所以再次通过nls2输入该对象以创建一个"nls"对象,允许我们使用fitted方法。我们绘制出相应的拟合图。
library(nlmrt)
library(nls2)
st <- c(Epsup=1, Bup=1, Epsdn=10, Bdn=-5)
fo <- y ~ (1/(1+sqrt(Epsup^2)/x)^Bup)*(1/(1+sqrt(Epsdn^2)/x)^Bdn)
fit.nls2 <- nls2(fo, start = data.frame(rbind(st/10, 10*st)), alg = "brute")
fit.nlxb <- nlxb(fo, data = data.frame(x, y), start = coef(fit.nls2))提供以下内容:
> fit.nlxb
nlmrt class object: x
residual sumsquares = 171.2 on 11 observations
after 19 Jacobian and 25 function evaluations
name coeff SE tstat pval gradient JSingval
Epsup 10.7464 10.95 0.9814 0.3591 6.855e-05 1584
Bup 1.15049 0.5928 1.941 0.09345 0.001839 120.2
Epsdn 642.754 908.5 0.7075 0.5021 -1.298e-06 1.406
Bdn -1.13885 0.6315 -1.804 0.1143 0.004964 0.005443 并绘制图解,以直观地评估适合性:
fit.nlxb.nls <- nls2(fo, start = coef(fit.nlxb))
plot(y ~ x)
lines(fitted(fit.nlxb.nls) ~ x)
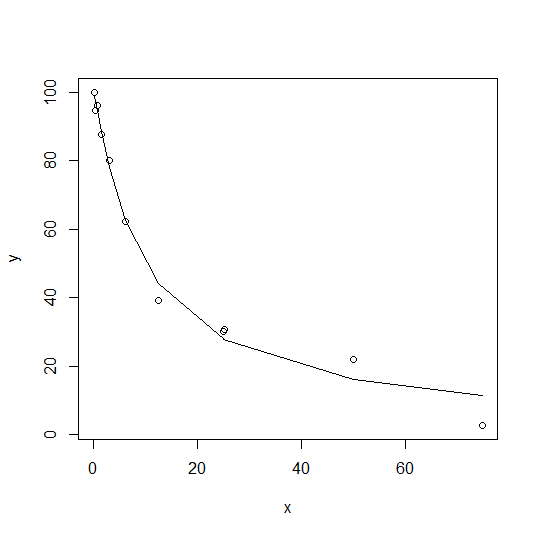
注意:我们使用了这个输入:
via_data <- data.frame(Concentration = c(0.197, 0.398, 0.792, 1.575,
3.154, 6.270, 12.625, 25.277, 25.110, 49.945, 74.680),
Viability = c(100, 94.62, 96.21, 87.53, 80, 62.22, 39.11,
30.80, 30, 22, 2.56))
x <- via_data$Concentration
y <- via_data$Viability页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/42511278
复制相关文章
相似问题














