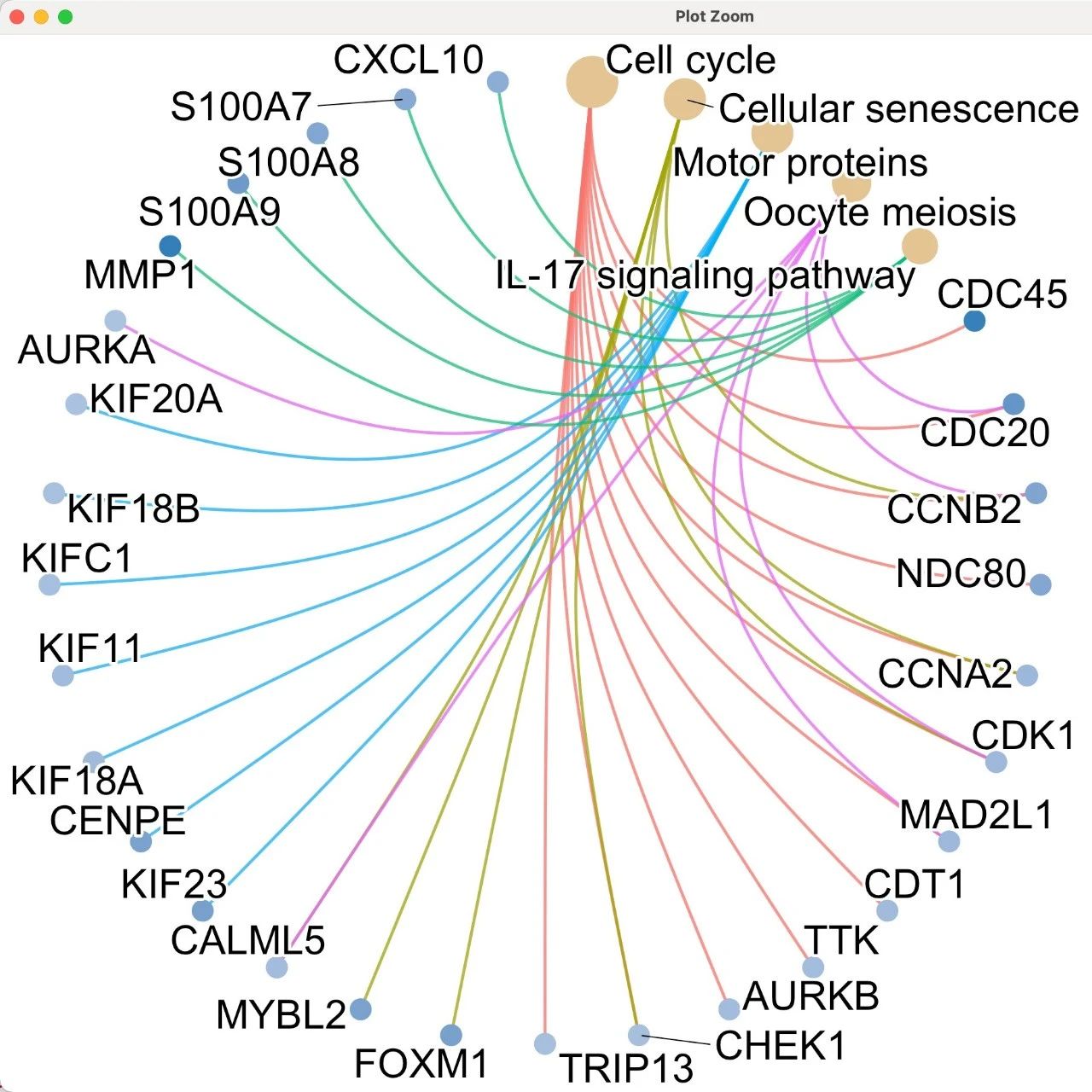
我有一个基因列表文件。像这样的事情
SWT21
SSA1
NRP1
EFB1
TFC3
MDM10我还有另外一个文件,其中也包含了这些基因的名字,以及关于它们的其他基本信息。第二个文件如下所示:
chrI 147593 151166 YAL001C - TFC3
chrI 143706 147531 YAL002W + VPS8
chrI 142173 143160 YAL003W + EFB1
chrI 140759 141407 YAL004W + YAL004W
chrI 139502 141431 YAL005C - SSA1
chrI 137697 138345 YAL007C - ERP2
chrI 136913 137510 YAL008W + FUN14
chrI 135853 136633 YAL009W + SPO7
chrI 134183 135665 YAL010C - MDM10我想从第二个文件中提取出那些在第一个文件中有基因名称的行。
回答 2
Unix & Linux用户
发布于 2014-03-24 12:36:56
您所需要的只是一个简单的grep:
grep -Fwf gene_list.txt gene_info.txt所采用的备选方案如下:
- -w :搜索整个单词,这将确保基因名
ERK1与基因ERK12不匹配(-w不是标准选项,但相当常见)。 - -f :从文件中读取要搜索的模式。在这种情况下,
gene_list.txt。 - -F :将模式视为字符串,而不是正则表达式。这确保了像
TOR*这样的基因名称(如果存在这样的东西)将与TORRRRRR不匹配。
注意:这假设列表中的基因名称周围没有空格。如果存在,则需要首先删除它们(在这里使用GNU sed):
sed -i 's/ //g' gene_list.txtUnix & Linux用户
发布于 2022-05-22 04:46:38
您还可以在linux中使用grep命令,方法是发出命令,如egrep -wi“完整的基因列表(1)与基因间的诱导管道”,文件名包含所有基因(2)“.Here genelist(1)是您的基因学家说它的数目为20,而geneliust (2)包含有整个基因的文件(1000 )”。
页面原文内容由Unix & Linux提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://unix.stackexchange.com/questions/121222
复制相关文章