开源数据代码,学习如何通过单细胞多组学数据鉴定关键因子
开源数据代码,学习如何通过单细胞多组学数据鉴定关键因子

Basic Information
- 英文标题:Multiomic single-cell profiling identifies critical regulators of postnatal brain
- 中文标题:多组学单细胞分析鉴定出调控产后大脑发育的关键因子
- 发表日期:17 February 2025
- 文章类型:Article
- 所属期刊:Nature Genetics
- 文章作者:Tereza Clarence | Panos Roussos
- 文章链接:https://www.nature.com/articles/s41588-025-02083-8
Abstract
Para_01
- 人类大脑的发育从胚胎发生开始,持续到成年期,其动态基因表达受细胞类型特异性顺式调控元件活性和三维基因组组织的控制。
- 为了加深对出生后大脑发育的理解,我们同时分析了来自十名捐赠者的四个脑区的101,924个单核的基因表达和染色质可及性,覆盖了从婴儿期到晚年的五个关键出生后期阶段。
- 利用该数据集和染色体构象捕获数据,我们构建了基于增强子的基因调控网络,以识别特定细胞类型的脑发育调节因子,并解释针对十种主要脑疾病的全基因组关联研究位点。
- 我们的分析将2,318个细胞特异性位点连接到1,149个独特基因,占所研究特征相关位点的41%,并突出了55个影响多种疾病表型的基因。
- 伪时间分析揭示了出生后少突胶质细胞生成的不同阶段及其调控程序。
- 这些发现为出生后大脑发育关键时间点的细胞类型特异性基因调控提供了全面的数据集。
Main
Para_01
- 成年人大脑由数十亿功能多样的细胞组成,是一个动态的生态系统,在早期发育过程中从神经外胚层中出现,并形成功能连接的结构。
- 这一过程伴随着神经发生和胶质发生的过程,这些过程协调了不同脑区中特定细胞类型的形成。
- 在分子水平上,已提出关键的基因调控程序来指导脑区的发育及相关细胞类型的特化。
- 尽管在理解这些过程方面取得了显著进展,但我们对个体细胞如何在出生后分化、获得功能性成熟以及经历与年龄相关的改变的理解仍然有限。
Para_02
- 细胞类型身份的成熟和维持,如基因表达水平所反映的那样,由特定转录因子(TFs)结合到顺式调控DNA元件(CREs)来控制,从而调节细胞的转录组状态。
- 鉴于其与精神疾病和神经退行性疾病风险的关联,人们投入了大量努力来解析人类产后大脑发育和自然衰老的表观基因组调控机制。
- 单细胞技术的进步为了解大脑发育提供了见解,但同时涵盖基因表达和染色质可及性的多组学分析仅在少数研究中得以应用。
- 此前,这种方法识别出了协调胚胎期人类大脑发育过程中基因表达和神经元谱系转换的转录因子——这一过程主要由神经发生驱动,表现出动态的细胞变化。
- 相比之下,产后大脑发育涉及不同的过程,例如突触修剪、髓鞘化以及神经回路的持续成熟。
- 值得注意的是,我们的分析重点在于产后少突胶质细胞生成,即新少突胶质细胞(OL)的产生,这对髓鞘化和长期神经功能至关重要。
Para_03
- 为了加深我们对支撑人类产后大脑发育的调控程序的理解,我们同时分析了从十个个体中分离出的单个细胞核中的基因表达和染色质可及性,这些个体涵盖了五个产后时间点(婴儿期、童年中期、青春期、成年早期和成年晚期),并涉及四个功能不同的脑区(前扣带回皮层 (ACC)、背外侧前额叶皮层 (DLPFC)、海马 (Hipp) 和尾状核 (CN))。
- 利用增强子驱动的基因调控网络(eGRNs),我们识别出了细胞类型特异性的增强子驱动调控因子集合(eRegulons),特别关注了产后的少突胶质细胞生成过程。
- 此外,通过与同一队列中细胞类型和发育阶段特异性的染色质接触图谱数据集整合,我们重建了一个细胞类型水平的染色质峰-基因关联图谱,以优先筛选之前与主要脑相关疾病相关的遗传位点的候选效应基因。
- 我们的研究结果构成了一个广泛的数据库,展示了在产后发育的关键时间点上调控谱系决定的细胞类型特异性机制,为理解支撑人类产后大脑发育的调控程序提供了深入见解,并确定了十种主要脑相关疾病的候选因果基因。
Results
Multiome profiling insinuated dominating temporal diversity
多组学分析暗示了主导的时间多样性
Para_01
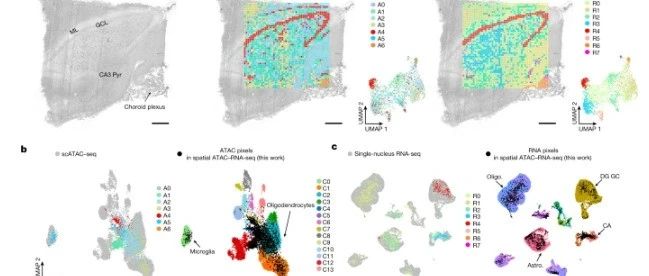
- 我们同时分析了从四个不同脑区(ACC、DLPFC、CN、Hipp)分离的单个细胞核中的转录组和染色质可及性,这些样本来自五个不同的发育阶段(婴儿期(0岁)、童年中期(4-6岁)、青春期(14岁)、成年早期(20-39岁)和成年晚期(61-62岁))(每个时间点2名个体)(图1a)。
- 总计101,924个细胞核通过了RNA测序(RNA-seq)和ATAC测序(使用测序技术检测转座酶可及染色质的实验方法)的严格过滤和质量控制标准(扩展数据图1a和2a-c,补充图1和补充数据1;方法)。
Fig. 1: Multimodal profiling and multivariate exploration of the postnatal human brain.
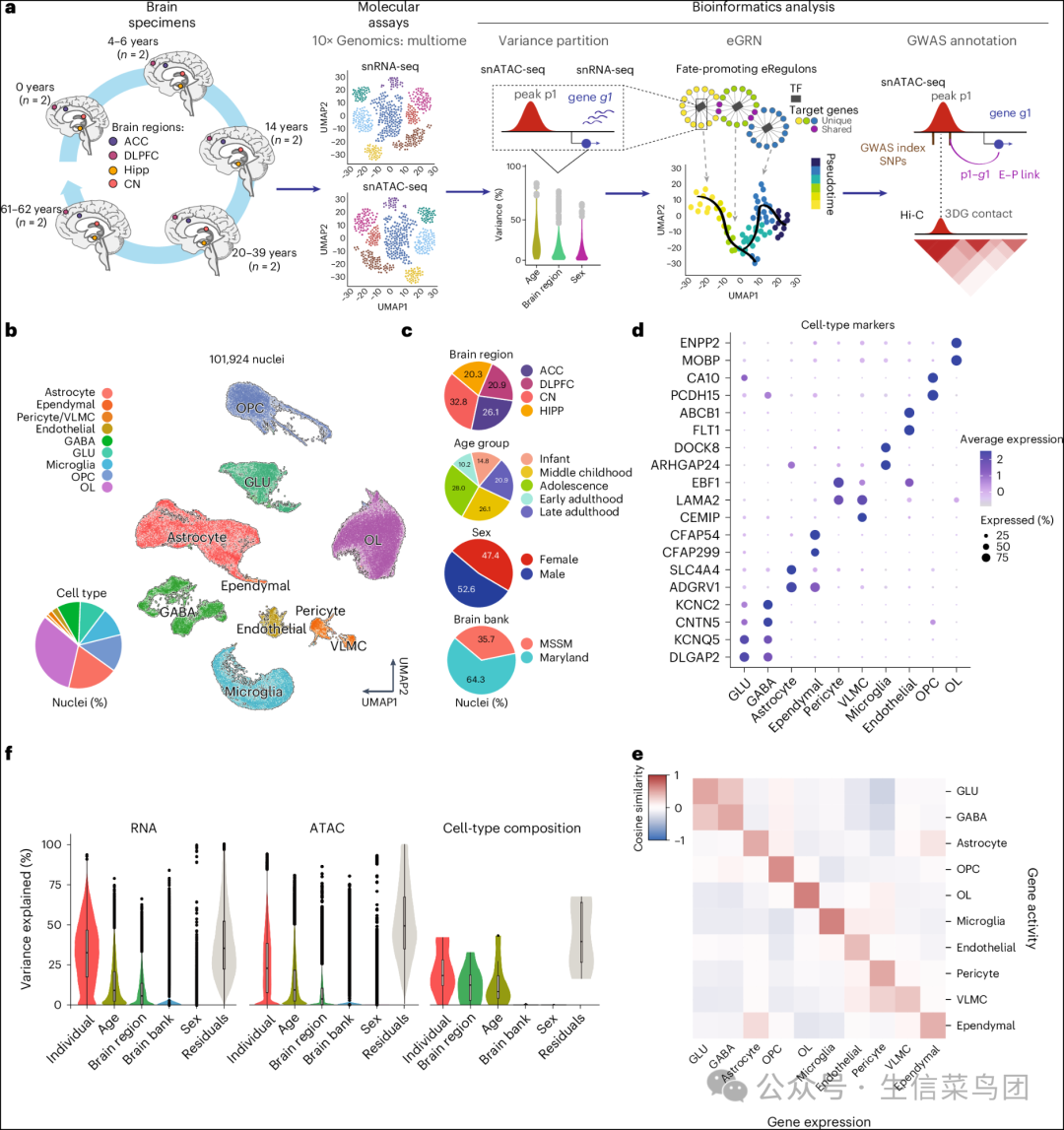
- 图片说明
◉ 研究设计和计算分析的示意图。3DG,三维基因组。◉ WNN 表示的单核 RNA 测序和单核 ATAC 测序数据投影到 UMAP 上,显示主要细胞类型。◉ 伴随的饼图展示了在主要协变量(性别、脑库、脑区、年龄组和细胞类型)中代表的细胞核比例。MSSM,西奈山医学院。◉ 基于最高平均表达量的前两个基因标记可视化其在主要细胞类型中的基因表达。◉ 主要细胞类型中基因表达与相应基因活性(由染色质可及性推导)之间的余弦相似度。◉ 使用方差分解方法分析协变量对基因表达(左,n = 16,661 基因)、染色质可及性(中,n = 181,432 峰值)和细胞类型组成(右,n = 10 种细胞类型)的解释方差。在每个小提琴图中,中心线表示中位数,箱子表示四分位距(IQR),而须线表示在 1.5× IQR 范围内的最高/最低值。超出第一和第三四分位数 1.5 倍 IQR 的点被视为异常值。
Para_02
- 此外,为了构建增强子-启动子(E-P)连接,我们利用了来自相同样本和脑区的荧光激活核分选(FANS)、细胞类型特异性和发育阶段特异性的Hi-C数据。
- 通过FANS分离出少突胶质细胞(OL)、γ-氨基丁酸能神经元(GABA)、谷氨酸能神经元(GLU)的细胞核以及由小胶质细胞和星形胶质细胞组成的混合群体(MG/AS)(扩展数据图1b-d;方法),并进一步处理以生成Hi-C文库(方法)。
Para_03
- 利用单核多组学数据,我们总共识别出十种细胞类型(星形胶质细胞、小胶质细胞、谷氨酸能和伽马氨基丁酸能神经元、内皮细胞、周细胞和血管性软脑膜细胞、室管膜细胞、少突胶质前体细胞以及少突胶质细胞)。
- 我们恢复了细胞类型特异性的标记,并从染色质可及性推断了其相应的基因活性,同时观察到基因表达谱与基因活性之间存在强烈的细胞类型特异性对应关系,尤其是在神经元细胞中,这与之前的文献一致。
- 我们使用多个已发表的数据集验证了我们分类的稳健性,并计算了参考数据集中细胞类型与我们细胞类型分类之间的成对余弦相似度。
Para_04
- 由于我们的数据集具有多维特性,涵盖了不同的发育阶段、脑区、脑库和捐赠者,我们在进行进一步的下游分析之前,首先对变异来源进行了分解。
- 我们对基因表达、染色质可及性和细胞类型组成进行了方差分解分析,估计了每个协变量所能解释的方差比例。
- 在考虑个体间变异后,基因表达和染色质可及性的变异主要由年龄驱动,其次是脑区(图1f)。
- 相比之下,细胞类型组成的变异主要受脑区特异性影响,其次是年龄(图1f)。
- 这与之前关于人脑细胞类型组成变化的报道一致。
- 由年龄驱动变异最大的前500个基因涉及中枢神经系统和器官发育相关的生物过程,而由脑区驱动变异的基因则参与脑发育、突触形成和传递(扩展数据图2d)。
- 有趣的是,在受性别影响最大的基因中,组蛋白甲基化富集。
eGRN identifies key postnatal regulators
eGRN 识别关键的产后调节因子
Para_01
- 为了进一步利用我们的多模态数据集的力量并确定调控细胞类型特异性的调控程序,我们通过整合增强子区域与候选上游转录因子(TFs),并将它们与下游靶基因相连,利用SCENIC+框架推断出增强子调控网络(eGRNs)。
- 这种方法能够检测所谓的eRegulons——由共同转录因子调控的一组基因及其相关的增强子(图2a)。
- 对eRegulon富集分数进行降维分析,区分了主要的细胞状态,同时重现了先前确定的细胞类型身份。
- 在应用过滤标准后(方法部分),eGRN分析鉴定了146个激活型eRegulons。
- 这些由增强子驱动的调控单元包含78,936个增强子区域,并与14,064个靶基因相关联。
- 在分析的基因中,81.8%的基因具有一个到十个预测的增强子,其中所有预测增强子中有74.7%被认为主要调控最近的基因。
Fig. 2: Cell-type level eGRN analysis in postnatal human brain.
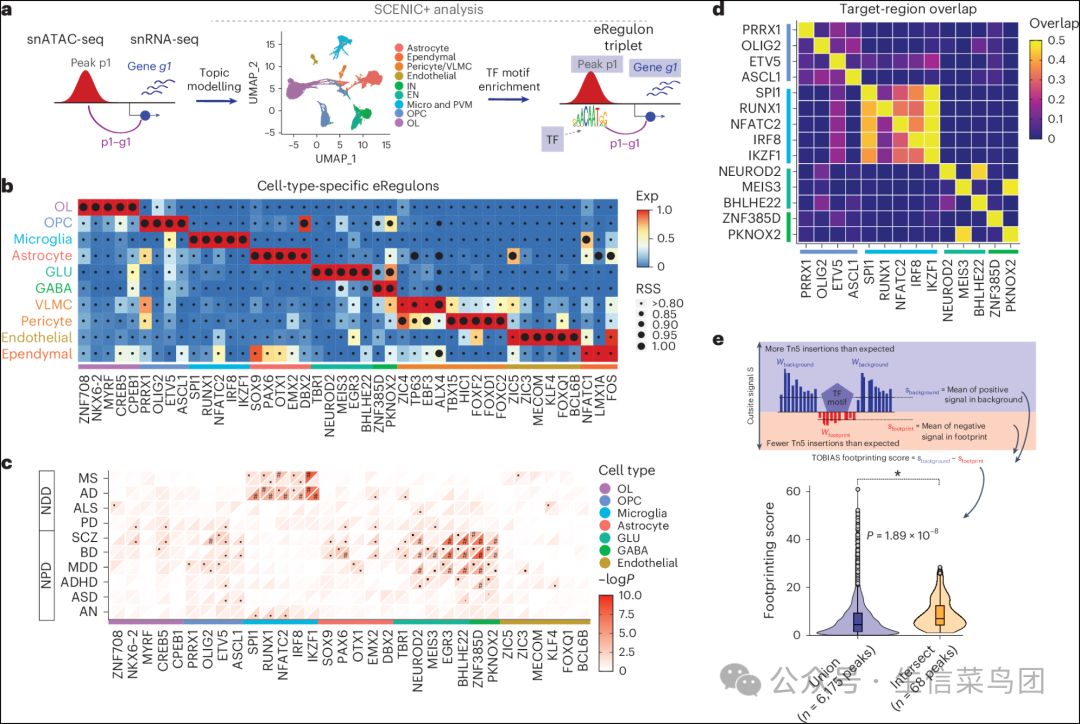
- 图片说明
◉ a,eRegulon检测方法的示意图。◉ b,热图/点图显示每种细胞类型基于其RSS特异性评分的三到五个最特异的eRegulons。缩放后的eRegulon表达以颜色表示,RSS以大小比例显示。◉ c,与选定疾病特征相关的常见风险变异在受细胞特异性转录因子调控的基因中的遗传力富集(左上三角;通过MAGMA计算)以及这些基因的增强子中的遗传力富集(右下三角;通过S-LDSC计算)。MAGMA富集分析显示了b中每种细胞类型调控因子对应的基因在选定性状中的富集情况。井号表示在所有测试的FDR(Benjamini-Hochberg校正)多重检验后P值为0.05时MAGMA中的显著富集;小黑方块表示在P值为0.05时显著富集。NPD,神经精神疾病;NDD,神经退行性疾病。◉ d,b中选定调控因子对之间共享目标区域的保守性。成对方式显示共享峰的比例,黄色对应最高值。◉ e,使用TOBIAS包计算足迹评分的概念化说明(顶部)。比较微胶质细胞TF基序SPI1、RUNX1、NFATC2、IRF8和IKZF1在eGRN分析识别为它们共享共靶标的峰上的足迹评分(橙色小提琴图,n=68)与仅识别为其中一个靶标的峰上的足迹评分(紫色小提琴图,n=6,175)(底部)。两组之间的差异通过单侧Kolmogorov-Smirnov检验在P值<10^-8时具有显著性。在每个小提琴图中,中心线表示中位数,盒子显示四分位距,须线表示在1.5倍四分位距范围内的最高/最低值。足迹图示源自原论文并经作者许可使用。
Para_02
- 我们进一步根据调控网络特异性评分(RSS)过滤了eRegulons,该评分在SCENIC+管道中计算,用于评估一个调控网络与特定细胞类型的相关性,通过比较其在多种细胞类型中的表达实现。
- 然后我们选择了排名前三到五位的最具有细胞类型特异性的调控因子(图2b)。
- 这些增强子调控网络(eGRNs)成功恢复了已知的少突胶质细胞(OL)和少突胶质前体细胞(OPC)的调控因子(分别为MYRF、CREB5和ASCL1,以及OLIG2)、星形胶质细胞(PAX6、SOX9)、小胶质细胞(SPI1、IRF8、RUNX1)、神经元(MEIS3和NEUROD2)、内皮细胞和周细胞/血管平滑肌细胞(FOX和ZIC转录因子家族)的调控因子(图2b)。
- 鉴于我们的数据集包含时间维度,我们探索了主要年龄段间调控因子的活性,并观察到了显著波动。
- 在大多数情况下,eRegulon的活性在不同年龄段表现出动态波动(补充图3)。
- OPC特异性的eRegulons从婴儿期到青春期表现出强大的特异性和表达水平,而神经元特异性的eRegulons则表现出相反的趋势,其特异性和表达水平在成年期逐渐增加(补充图3)。
Para_03
- 我们进一步通过 MAGMA 和 S-LDSC 分析方法,分别基于基因表达和染色质可及性,表征了细胞类型特异性调控因子的疾病相关风险。
- 小胶质细胞特异性调控因子与阿尔茨海默病(AD)和多发性硬化症(MS)表现出强烈的关联(图 2c 和补充数据 4)。
- 少突胶质前体细胞(OPC)特异性调控因子与精神分裂症(SCZ)相关,而神经元调控因子则与神经精神特征相关,例如重度抑郁症(MDD)、双相情感障碍(BP)和注意缺陷多动障碍(ADHD)。
- 将这种方法扩展到所有 eRegulons,我们全面优先级化了每个调控因子与特定疾病和细胞类型的关联(扩展数据图 3 和补充数据 5)。
Para_04
- 接下来,我们寻找调控因子之间的关系,并检查了它们的增强子和目标基因的重叠情况(图2d和扩展数据图2d)。
- 小胶质细胞特异性调控因子在增强子区域和目标基因中表现出最高的重叠度。
- 我们假设共享目标基因的转录因子(TFs)也会在增强子区域中表现出富集的足迹,这支持了它们潜在的共调控作用。
- 通过使用TOBIAS工具进行转录因子足迹分析,进一步验证了这一假设。
- 我们观察到,在共享增强子区域的基序上,小胶质细胞特异性调控因子(SPI1/RUNX1/NFATC2/IRF8/IKZF1)的结合强度显著高于仅与单个调控因子相关的增强子区域(图2e)。
Enhancer–gene networks reveal risk loci for brain disorders
增强子-基因网络揭示了大脑疾病的风险位点
Para_01
- 标注特定细胞类型在大脑相关特征中的作用仍然很大程度上不完整,特别是考虑到超过90%的疾病位点位于非编码区域,并且高质量的组织和细胞特异性数据(涵盖多种组学模式)仍然缺乏。
- 通过使用连锁不平衡(LD)评分回归(LDSC)方法(方法部分),分析大脑相关的全基因组关联研究(GWAS)以及细胞类型特异性的基因和染色质峰的遗传力富集情况,我们分别发现了22个和19个显著关联(Benjamini–Hochberg校正后的P值<0.05)(图3a和补充数据7)。
- 值得注意的是,许多关联在这两种分析中都被发现(28对细胞-疾病对中有14对是共享的;Spearman相关系数ρ=0.65),这表明每种分析都能捕捉到可靠和有意义的关联。
- 测量到的富集情况大多与现有的细胞类型-特征关联相一致(扩展数据图4)。
- 前额叶皮层中GABA能和谷氨酸能神经递质功能障碍是许多精神疾病共有的特征,包括精神分裂症、双相情感障碍、重度抑郁症、注意缺陷多动障碍和自闭症谱系障碍。
- 此外,越来越多的证据表明,少突胶质细胞和少突胶质前体细胞在这些精神疾病中对调节神经元间通讯的有效性和精确性起着重要作用。
- 相比之下,阿尔茨海默病、帕金森病和多发性硬化症则主要通过与慢性免疫激活和神经炎症相关的机制牵涉小胶质细胞。
Fig. 3: Summary of cell-type-specific disease heritability and disease regulome landscape.

- 图片说明
◉ 通过细胞特异性标志基因(RNA-seq)和峰(ATAC-seq)计算脑相关疾病中脑细胞类型的遗传力富集。垂直虚线表示名义显著性的阈值(RNA-seq MAGMA/ATAC-seq LDSR 回归 P 值为 0.05)。点的大小表示在应用 Benjamini–Hochberg 方法控制 FDR 在 0.05 后的统计显著性。NS,不显著。◉ 根据峰所具有的至少一个 E–P ABC 链接的细胞类型数量,对峰进行分组后,突变约束 Z 分数的平均值。Z 分数来源于全基因组突变约束图。◉ 每个 GWAS 特征在所有研究的细胞类型中解释的位点的相对比例(左 y 轴,柱状图)和绝对数量(右 y 轴,实线)。◉ GWAS 风险变异与假设调控基因转录起始位点(TSS)之间的 E–PABC-MAX 距离直方图,其中红色和蓝色虚线分别表示中位数和平均值。◉ 达到假设调控基因时被 E–PABC-MAX ‘跳过’ 的基因数量的直方图,其中红色和蓝色虚线分别表示中位数和平均值。◉ 在所有研究的 GWAS 特征中,每种细胞类型解释的位点数量。
Para_02
- 虽然确定细胞类型富集有助于揭示疾病遗传力的一般模式,但我们的目标是通过以细胞类型特异性方式提名与疾病相关的候选基因来深入研究。
- 为此,我们使用 ABC 方法将与疾病相关的位点与候选效应基因联系起来,利用来自七个主要脑细胞类型的年龄组和细胞特异性 Hi-C 数据中的 E-P 相互作用(补充图 4a)。
- 包含 272 种细胞类型、脑区和供体特异性的调控组图谱的综合阵列改进了 eGRN 分析,通过为 E-P 相互作用提供直接的功能证据而优于传统方法。
Para_03
- 总体而言,我们鉴定了19,500个表达基因和273,815个独特的染色质可及性峰之间的4,707,778个增强子-启动子(E–P)链接。
- 不同细胞类型之间E–P链接的数量差异显著,其中谷氨酸能神经元的链接数量最高,反映了这些细胞中远端基因间调控复杂性的差异。
- 平均而言,21%的E–P链接在至少两种细胞类型中共享,而在相同细胞类型内跨脑区分析时,29%至52%的E–P链接是共享的。
- 与最近的一项全基因组突变约束图谱研究结果一致,该研究评估了整个基因组中遗传变异的缺失情况,我们的发现证实参与E–P相互作用的峰值受到更强的负向选择,而非预测为不参与此类相互作用的峰值。
- 给定峰值所涉及的调控相互作用的细胞类型数量越多,其负向选择的强度越高。
Para_04
- 接下来,我们旨在利用增强子-启动子(E-P)连接为全基因组关联研究(GWAS)确定的疾病相关位点提名候选的功能基因(补充图4a)。
- 首先,我们收集了一组与选定神经精神和神经退行性特征相关的1,156个全基因组显著变异(P < 5 × 10⁻⁸),并将该集合扩展到包括30,694个基于高连锁不平衡(LD,R² ≥ 0.8)的变异。
- 然后,我们对这些潜在与疾病相关的变异和细胞特异性E-P连接进行了重叠分析(方法)。
- 为了避免一个位点出现多个关联,我们采用了ABC-MAX方法,并对每个峰仅保留具有最高ABC评分的E-PABC连接(E-PABC-MAX)(补充数据8)。
- 在所有细胞类型中,我们总共提名了1,149个独特的基因,这些基因可能与所研究的GWAS特征中41%的位点相关(图3c,补充数据9和补充图5-10)。
- E-PABC-MAX的距离差异显著,尽管其中大多数(61%)距离小于100 kb(中位数为53 kb)(图3d)。
- 尽管如此,绝大多数(66%)的E-PABC-MAX连接并未指向最近的基因(到最近基因的中位距离为7.8 kb),其中15%跨越了十个或更多基因(图3e和补充注释1)。
Para_05
- 基于我们之前的研究发现,即在神经元亚型中,所研究的脑相关疾病显示出最强的遗传力信号(图3a),我们进一步通过确认谷氨酸能和γ-氨基丁酸能神经元中最多的E–PABC-MAX数量验证了这一趋势(图3f)。
- 与已知的疾病生物学一致,小胶质细胞在神经退行性疾病中特异性富集,而在精神疾病中则不然,其中阿尔茨海默病、多发性硬化症显示出最高的E–PABC-MAX数量,并且小胶质细胞是帕金森病中富集程度最高的细胞类型之一(补充数据9)。
Prioritized enhancer–gene networks identify causal genes in selected brain disorders
优先级增强子-基因网络确定了选定脑部疾病中的致病基因
Para_01
- 这些E-P预测为识别GWAS位点的功能基因、通路和调控特性提供了资源。
- 我们的方法对与多发性硬化症(MS)相关的85个位点进行了预测,提名了总计164个独特基因。
- 其中许多基因之前已被证实与针对中枢神经系统(CNS)的自身免疫炎症过程有关,并导致神经退行性病变。
- 值得注意的是,最显著的富集出现在通过小胶质细胞E-PABC-MAX连接关联的基因中。
- 这些基因积极参与细胞因子反应的调节(18个基因,比值比=8),α/β T细胞激活(10个基因,比值比=15),白介素-2受体信号传导(IL2RB;5个基因,比值比=48)以及白介素-15信号传导(IL-15;4个基因,比值比=89)。
- 在多发性硬化症的背景下,调控这些富集通路相关基因的增强子主要表现出小胶质细胞特异性。
- 这一点在白介素-2受体信号传导通路中尤为明显,因为已知小胶质细胞表达白介素-2和其他细胞因子的受体,从而能够调节T细胞介导的免疫反应。
Fig. 4: Annotation of GWAS prioritized genes.

- 图片说明
◉ a,基因集之间的重叠(通过单侧 Fisher 精确检验评估),表示生物学通路和在 MS GWAS 中每种细胞类型优先级的基因。显示了前十个交集,垂直虚线表示 P 值 0.05 的名义显著性(单侧 Fisher 精确检验),点的大小反映了 FDR(Benjamini-Hochberg 校正)显著性。仅包括具有 FDR 显著富集的细胞类型。◉ b,SynGO 数据库中突触层次结构内优先级基因的定位。日爆图以突触为中心,第一环为突触前和突触后位置,后续环为子术语。图例中的颜色方案表示每个术语中的基因数量。选择了部分功能术语和基因进行标记。◉ c,我们方法为 ALS GWAS 优先级确定的 16 个基因集合。‘先前证据’来源于 ALS GWAS。红色刻度反映了连接基因与 GWAS 位点的 E-PABC-MAX 链接的 ABC 分数。蓝色分数描绘了增强子与启动子之间的 log10(E-PABC-MAX 距离)。◉ d,归一化的 snATAC-seq 派生伪批量轨迹,展示了 C9orf72 基因复杂的细胞特异性调控,并突出显示了与 ALS GWAS 相关的小胶质细胞 E-P 链接。每个伪批量轨迹的 y 轴已归一化,对应信号强度(最大值为 120)。Chr 表示染色体。
Para_02
- 为了进一步证明我们方法的准确性,我们研究了137个SCZ位点(占所有SCZ位点的55%),这些位点具有预测的E–PABC-MAX链接(补充数据9)。
- 在将我们预测的因果基因与最新SCZ GWAS中通过SMR/FINEMAP优先级排序的基因进行比较时,我们发现两份研究共同覆盖的47个位点中有29个(62%)一致。
- 值得注意的是,在18个预测因果基因不同的位点中,有7个位点我们确认存在与SMR/FINEMAP预测相同基因的E–PABC链接(但不是E–PABC-MAX链接),这表明即使不是排名最高的链接,在我们的数据中仍然存在调控链接。
- 与全基因组富集测试结果一致,我们发现优先级较高的候选蛋白编码基因涉及突触前和突触后病理(图4b)。
- 这些优先级较高的基因与突触层次结构中的32个独特生物学术语相关(补充数据11),包括若干特定通路和过程。
- 例如,一些基因编码参与突触前和突触后元件之间黏附的蛋白质(EFNA5、PTPRD)、钙离子、氯离子和钾电压门控通道(CACNA1D、CLCN3和KCNB1),以及配体门控离子通道(GRIN2A、CHRNA3)(补充数据11)。
- 值得注意的是,GRIN2A编码N-甲基-D-天冬氨酸受体的一个亚基,并包含罕见和常见的SCZ风险变异。
- 其中一些罕见变异可能影响通道功能,大多数预计会导致蛋白质截短,最终导致基因表达降低,类似于常见变异(补充图12)。
Para_03
- 接下来,我们探讨了肌萎缩侧索硬化症(ALS)的精细定位结果——与多发性硬化症(MS)或精神分裂症(SCZ)相比,ALS 的全基因组关联研究(GWAS)中涉及的全基因组显著位点数量明显较少。
- 这种差异主要归因于 ALS 样本量较小以及其独特的遗传结构,表现为较低的多基因性特征。
- 我们的方法成功识别出 16 个位点中的 8 个潜在因果基因(图 4c)。
- 其中,七个基因被 E–PABC-MAX 和最新的 ALS GWAS 同时列为优先级较高的基因,包括 C9orf72 和 TBK1,这两个基因包含罕见和常见变异(图 4d 和补充图 13)。
- 值得注意的是,在 C9orf72 基因的情况下,导致疾病的变异是罕见的六核苷酸 (G4C2)n 重复扩展,而不是常见的指数变异 rs2453555,这才是 C9orf72 位点内的因果变异。
- 尽管 C9orf72 基因在大脑中广泛表达,但与指数变异 rs2453555 重叠的 E–PABC-MAX 调控区域仅在小胶质细胞中具有可及性(图 4d)。
- 这一发现进一步强调了外周髓系细胞,特别是小胶质细胞,在 ALS 病理生理学中的重要性。
Para_04
- 最后,我们的方法确定了一组与多种疾病相关的55个基因,其中显著的是叉头框蛋白P1(FOXP1)。
- FOXP1在神经性厌食症(AN)、注意力缺陷多动障碍(ADHD)、精神分裂症(SCZ)和多发性硬化症(MS)中的关联表明其在多样化的认知和社会过程中起着关键作用。
- 这种转录因子对大脑发育至关重要,长期与智力障碍及相关认知表型有关,主要通过新生变异和拷贝数变异实现。
- 最近针对AN、ADHD、SCZ和MS的大规模GWAS研究显示,在3p13位点上优先级较高的基因座存在显著重叠,一致将FOXP1确定为因果基因。
- 这种联系得到了表达数量性状位点(eQTL)数据的支持,并且体内实验表明,敲除FOXP1的小鼠体重减轻,证实了其与神经性厌食症的功能联系。
- 对于其与精神分裂症、多发性硬化症和注意力缺陷多动障碍的关联,也存在类似的有力证据。
- 总体而言,这些发现强调了FOXP1作为各种脑相关疾病共有病理生理学的核心节点的关键作用。
Pseudotime analysis defines key steps in oligodendrogenesis
伪时间分析定义了少突胶质细胞生成的关键步骤
Para_01
- 少突胶质细胞生成是一个特别有趣的过程,因为它代表了大脑中少数几个产后细胞分化的例子之一。
- 鉴于我们的数据集中少突胶质前体细胞(OPC)和少突胶质细胞(OL)群体的富集,我们进行了重新聚类以解析更精细的亚簇,识别出13个不同的亚簇(七个 OL 和六个 OPC)。
- 为了简化解释并增强生物学相关性,我们随后根据共享的基因表达谱和独特的标志物特征,将这些亚簇分组为常见的 OPC 和 OL 亚型(图5a和扩展数据图5a–c)。
Fig. 5: Characterization of oligodendrogenesis by integrative pseudotime analysis.

- 图片说明
◉ a,联合 ATAC-seq 和 RNA-seq(WNN)UMAP 表示的 OPC 和 OL 群体,按亚型注释着色。◉ b,堆叠条形图表示不同年龄组中 OPC 和 OL 亚型的比例,以及使用 crumblr(方法)计算的亚型组成随年龄的变化情况。x 轴表示效应大小,颜色和点的大小表示 log10(FDR);加号符号表示具有统计学显著性的结果;误差棒表示从标准误差计算出的 95% 置信区间。◉ c,monocle3 计算的伪时间值投影到 RNA-UMAP 上。◉ d,OPC 和 OL 亚型(左)及年龄组(右)的伪时间分布箱线图(左:n_OPC Type I = 12,445 核,n_OPC Type II = 1,371 核,n_OL Type I = 29,560 核,n_OL Type II = 3,691 核;右:n_infant = 5,153 核,n_childhood = 14,844 核,n_adolescence = 11,873 核,n_early_adulthood = 3,164 核,n_late_adulthood = 11,749 核)。中心线表示中位数,箱子表示四分位距(IQR),须表示在 1.5 倍 IQR 内的最大值和最小值。◉ e,通过层次聚类将热点基因模块分类为三种主要的基因表达趋势(补充图 17b,c)。上升趋势(红色)、下降趋势(绿色)和恒定趋势(灰色)。◉ f,每种趋势线的基因集富集分析(GSEA),显示生物过程中最富集的 Gene Ontology(GO)术语,点的大小表示基因数量,颜色表示调整后的 P 值(FDR,Benjamini–Hochberg 校正)(补充数据 15)。ER,内质网;SRP,信号识别颗粒。
Para_02
- 这导致发现了两类主要的 OPC,具体来说,I 型 OPC 表达 PDGFRA、GPC5、SNTG1、ATRNL1 和 NXPH1(与神经元信号传导相关),而 II 型 OPC 则显著表达 BCAS1、GPR17、BMPER 和 FRMD4A(补充数据 13),并与神经发育和神经退行性疾病存在潜在联系。
- 这两种亚型之前已经在人类和小鼠研究中被报道过。
- 此外,基于 OPALIN 和 GPR37(标记 I 型)以及 RBFOX1 和 ACTN2(II 型)的表达,还识别出了两种不同的少突胶质细胞(OL),这一结果与近期的研究报告一致。
- 所有 OPC 和 OL 亚型都在皮层(ACC、DLPFC)和皮层下(CN、Hipp)区域被检测到(扩展数据图 5b)。
Para_03
- 为了验证我们在单核RNA测序(snRNA-seq)研究中关于OPC和OL群体中同时存在I型和II型亚型的发现,我们进行了RNAscope实验。
- 为了可视化不同的OPC和OL亚型,我们集中研究表达特定标志物的细胞:PDGFRA、ATRNL1和BMPER(指示OPC亚型)以及GPR37、RBFOX1和ACTN2(指示OL亚型)(扩展数据图6a–b)。
- 与我们的计算分析一致,我们识别出了表达PDGFRA和ATRNL1的独特亚群,标记为I型OPC(扩展数据图6c–d),以及表达BMPER的另一亚群,标记为II型OPC(扩展数据图6c,e)。
- 同样,对于OL亚型,我们确认了不同的细胞亚群;表达GPR37的细胞标记为I型OL(扩展数据图6f,g),以及表达RBFOX1和ACTN2的细胞,标记为II型OL(扩展数据图6f,h)。
Para_04
- 总体而言,我们在婴儿期后观察到 OL 比例增加,而成年后 OPC 比例相应减少(图 5b)。
- 为了验证 OPC/OL 亚型比例随年龄变化的显著程度,我们使用 crumblr 进行了细胞类型组成分析(方法)。
- OPC Type II(GPR17、BMPER 富集)显示出最大的与年龄相关的效应大小,而 Type I(PDGFRA 富集)则具有边缘值,表明其在整个生命周期中的重要性。
- 随着年龄的增长,OL 的富集尤其体现在 OL Type I(OPALIN、GPR37 富集),而 Type II(RBFOX1 富集)仅表现出轻微的变化。
- 为了更深入地了解少突胶质细胞生成过程,我们进行了伪时间分析(图 5c 和扩展数据图 5c,d),该分析重现了之前从细胞亚型比例中观察到的结果:Type I 和 Type II OPC 富集在伪时间最低的细胞中。
- Type I OL(OPALIN 富集)似乎在整个伪时间过程中逐渐发展,并在后期表现出更显著的富集;而 Type II OL(RBFOX1 富集)在婴儿期几乎不存在,主要在生命的后期阶段开始出现(图 5b-d)。
- 这一趋势通过几种独立的方法(monocle3、Palantir 和 PC1)得到了高度相关性的验证(扩展数据图 5d-f)。
Para_05
- 为了在功能背景下整合 OPC 和 OL 亚型的形成,进行了热点模块分析。
- 这确定了 12 个不同的热点基因模块,涵盖了各种生物过程。
- 为了了解少突胶质细胞生成中的功能关联,我们接下来检查了单个热点模块沿伪时间的基因表达模式,并根据它们的相似性对它们进行了聚类。
- 这揭示了三条主要趋势线:上升(热点模块 1、3、8),下降(热点模块 2、4、5、6、7、11、12)和恒定(热点模块 9、10)。
- 在探索功能关联时,下降趋势线显示在化学突触传递和神经元投射等过程中富集,这与 OPC 活动相关。
- 相反,上升趋势线在脂质代谢过程中表现出富集,这与 OL 形成一致。
- 恒定趋势线与基本的管家生物过程相关。
Distinct eGRN activation reveals programs in oligodendrogenesis
不同的eGRN激活揭示了少突胶质细胞生成的程序
Para_01
- 在证明了某些 eRegulons 的活性与发育过程中的特定时间点相关联后,我们进一步试图通过伪时间与之前定义的 146 个激活型 eRegulons 活性评分的相关性分析,来识别维持少突胶质细胞生成的调控程序。
- 我们选择了与伪时间正相关和负相关的前十个 eRegulons(图 6a),这些 eRegulons 的基因表达模式与其对应的转录因子相同。
- 此外,这些与伪时间相关的 eRegulons 每一组都具有显著的目标基因重叠,但在目标区域水平上并未保留,这表明可能存在不同的共调控关系。
- 为了区分与伪时间早期阶段和晚期阶段相关的 eRegulons,我们将它们分别命名为早期或晚期少突胶质细胞生成相关 eRegulons(OLa-eRegulons)。
Fig. 6: Identification of key regulatory programs in oligodendrogenesis.
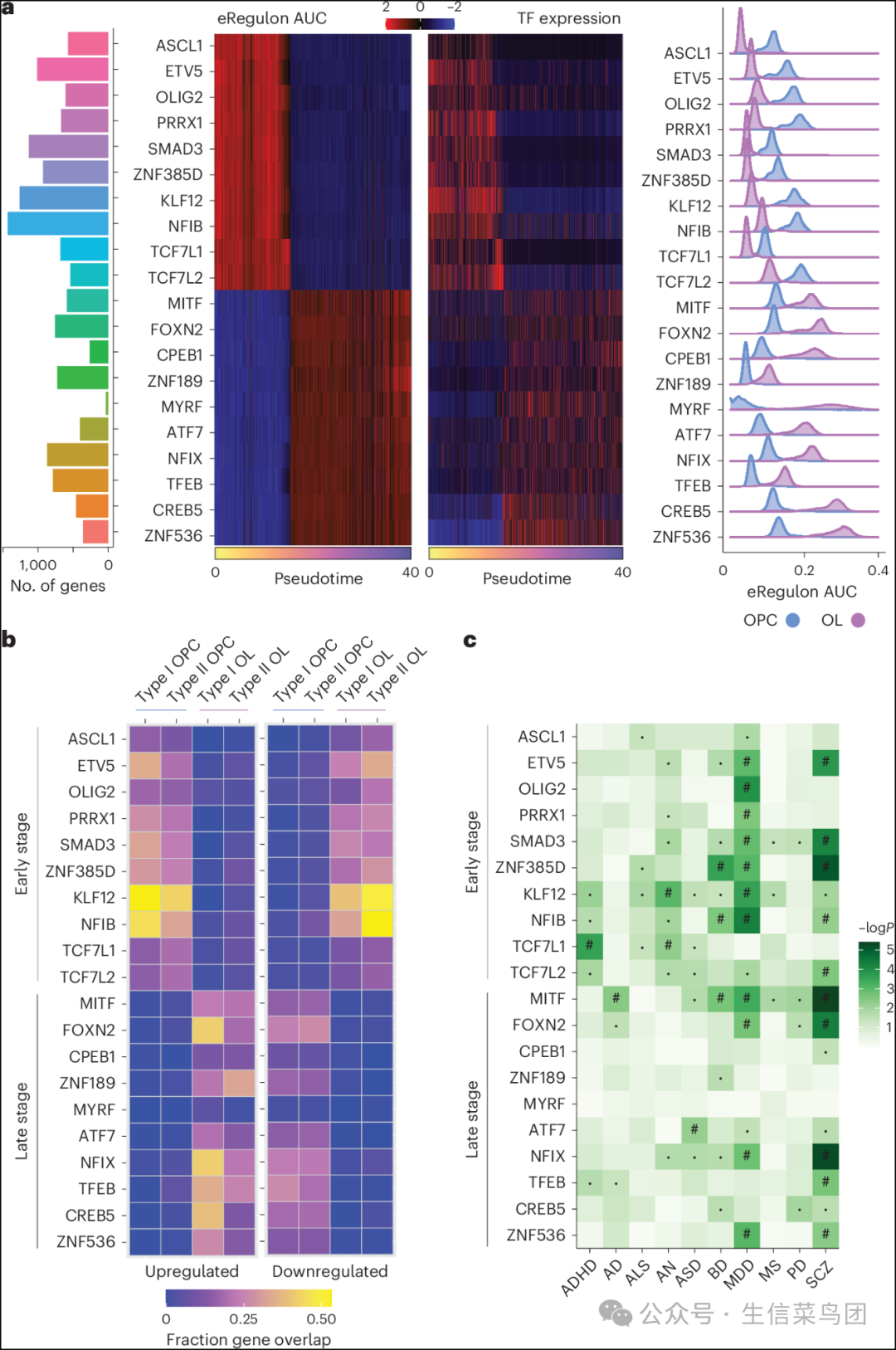
- 图片说明
◉ 排名前20的eRegulons与早期和晚期伪时间相关,条形图显示每个eRegulons中对应的基因数量(左侧)。◉ 沿少突胶质细胞生成伪时间的eRegulon AUCell评分和eRegulon转录因子表达值的热图来自图5c,侧边空白处指示相对表达水平(截断值-2,2)(中间)。◉ OPC和OL中OLa-eRegulons的AUCell评分分布。◉ OPC和OL亚型中上调和下调基因之间的相对成对重叠,与早期(浅灰色)和晚期(深灰色)阶段相关的eRegulons。◉ MAGMA富集分析针对十种与神经精神疾病和神经退行性疾病相关的特征,对早期和晚期OLa-eRegulons进行分析。带有井号标记的表示在MAGMA富集分析中P值<0.05具有显著性(经过所有测试的Benjamini-Hochberg校正多重检验);小黑方块表示在P值为0.05时具有显著性。
Para_02
- 在早期的 OLa-eRegulons 中,我们识别出了所有 OPC 特异性的调控因子(ETV5、ASCL1、OLIG2 和 PRRX1),以及更多的 OLa-eRegulons,包括 TCF/Lef 转录因子家族的两个成员、SMAD3、NFIB、KLF12 和 ZNF385D,其中 ZNF385D 被检测为 GABA 特异性的 eRegulon。
- TCFL1 和 TCFL2 因其在 WNT 信号通路中的作用而闻名,这些通路对调节 OPC 的增殖和分化至关重要。
- SMAD3 在中枢神经系统髓鞘化时间调控中起关键作用,作为转化生长因子 beta (TGFβ) 信号的介导者,调节 OPC 退出细胞周期。
- 有趣的是,NFIB(核因子-IB),lnc158 的目标转录本,已被报道显著促进少突胶质细胞(OL)分化。
- 而 KLF12 已被证明在大脑发育中发挥作用,可能通过神经元成熟的表观遗传转换实现这一功能,同时 ZNF385D 与阅读障碍和语言损伤有关,并且最近研究表明它可能在帕金森病(PD)进展中出现失调。
Para_03
- 在十大晚期少突胶质细胞(OL)特异性增强子调控网络中,我们鉴定出了CREB5和MYRF,它们是关键的少突胶质细胞特异性调控因子。
- 值得注意的是,ZNF536参与了对神经元分化的负向调控,而ZNF189则具有对抗应激诱导缺陷的保护作用,这两种因子被确定为重要的候选分子。
- 此外,ATF7与长寿相关,而NFIX则调控神经前体细胞的分化,这些转录因子在神经发育中的作用得到了强调。
- MiTF/TFE家族成员调控大脑的关键功能,例如自噬和线粒体稳态;在神经元中过表达TFEB显示出对帕金森病的保护作用,而在少突胶质细胞中它通过减轻多系统萎缩中的α-突触核蛋白积累发挥细胞特异性的神经保护作用,这表明其通过自噬-溶酶体途径具有细胞特异性的神经保护功能。
- FOXN2在少突胶质细胞中高表达,与大脑发育相关,并可能与语言和言语障碍有关。
Para_04
- AUCell(富集分数曲线下面积)的这些 OLa-eRegulons 的富集与 OPC 和 OL 亚型一致,早期阶段的 OLa-eRegulons 在 OPCs 中显著,而晚期阶段的 OLa-eRegulons 在 OL 中更为普遍。
- 此外,我们探索了 OPC 和 OL 亚型中上调和下调基因以及早期和晚期 OLa-eRegulons 目标基因之间的重叠情况。
- 我们发现,在 OPCs 中上调而在 OL 中下调的基因在早期阶段的 OLa-eRegulons 中显示出更高的重叠,尽管在 OPCs 中下调而在 OL 中上调的基因存在于晚期阶段的 OLa-eRegulons 中。
- 最后,我们通过 MAGMA 分析研究了早期和晚期 OLa-eRegulons 中调控基因与脑部疾病风险的关联性。
- 总体而言,早期阶段的 OLa-eRegulons 在 MDD 中显示出强烈的富集,除了 TCF7L1 外均显著,其次是 SCZ 显示出明显的富集。
- 相比之下,晚期阶段的 OLa-eRegulons 展现出更加多样化的富集模式,其中 MITF 在 AD、BD、MDD 和 SCZ 中均显著,而 FOXN2、CREB5 和 ZNF536 在 MDD 和 SCZ 中富集,其余晚期阶段的 OLa-eRegulons 显示出边缘或不显著的富集。
Discussion
Para_01
- 在这项研究中,我们构建了四个不同人类大脑区域(ACC、DLPFC、HIPP 和 CN)在五个出生后发育阶段的转录组和表观基因组多组学图谱。
- 通过使用协变量分析,我们发现大多数表达变异可归因于年龄,其次是大脑区域。
- 在染色质可及性变异中也观察到类似趋势,这表明我们的数据中基因表达与染色质可及性之间存在广泛联系。
- 此外,细胞类型组成的变化主要由大脑区域决定,同时也与年龄相关。
- 随后对 101,924 个单个细胞核的多模态分析识别出关键的增强子调控网络(eGRNs),并将共调控基因和顺式调控元件分类为特定细胞类型的单元。
- 预测的细胞类型特异性 eGRNs 的转录因子驱动因子与先前的文献一致。
Para_02
- 之前通过基因组距离和Hi-C衍生的染色质环成功推断了E-P接触。
- 为了改进这一点,我们采用了ABC方法来预测近500万个E-P链接的全基因组图谱,其中成千上万的链接控制着与脑部疾病相关的风险变异的表型表达。
- 这些图谱共同优先确定了1,149个候选效应基因,占分析位点的41%。
- 值得注意的是,对于大多数已确定有多个潜在目标基因的位点,此分析将候选基因缩小到单个基因,并将其分配给特定的细胞类型,从而提供了更多的生物学背景信息。
Para_03
- 通过整合之前由于生物样本有限而无法获得的大脑特异性图谱,我们的结果增强了现有的全组织努力,这些努力已经用于在72种疾病中绘制超过一百种人类细胞类型。
- 在我们的研究中,我们通过利用研究队列中所有个体生成的细胞和大脑区域特异性的Hi-C数据,进一步优化了E–P预测。
- 尽管我们的方法确实存在一个内在局限性,即假设每个疾病变异只有一个因果基因,这可能会忽略高度多效性效应,但我们认为这是一个合理的权衡。
- 这种方法使我们能够在细胞特异性背景下显著缩小与大脑相关疾病因果基因的搜索范围。
- 因此,我们预计我们的图谱将大大加速未来大脑相关研究中的变异到功能分析。
Para_04
- 我们对少突胶质细胞生成的研究揭示了具有特定转录组特征的不同OPC和OL群体。
- 值得注意的是,富集GPR17和BMPER的II型OPC在OL髓鞘形成的早期阶段中起着关键作用。
- GPR17在OPC分化过程中受WNT通路调控,在大脑发育期间充当重要的时间机制,而这两条通路的失调可能在如多发性硬化症等病理中具有重要意义。
- 此外,I型OL中富集的RBFOX1基因,其常见和罕见遗传变异均与广泛的精神疾病有关。
Para_05
- 基于这些见解,我们通过将 AUCell 分数与少突胶质前体细胞 (OPC) 和少突胶质细胞 (OL) 成熟的伪时间相关联,优先考虑调控少突胶质细胞发生关键阶段的核心 eRegulators,并鉴定出早期和晚期的 OLa-eRegulons。
- 这些转录因子 (TFs) 之前已被证明与早期 OLa-eRegulons 的祖细胞发育和细胞命运决策有关,而晚期 OLa-eRegulon 的 TFs 则被报道在神经元祖细胞分化和髓鞘形成的协调中发挥作用。
- 鉴定出的 OLa-eRegulons 同时调控许多与 OPC 和 OL 相关的标志物,表明这些转录因子是调控少突胶质细胞发生的至关重要的候选者。
- 同时,它们还对与精神疾病(如抑郁症和精神分裂症)相关的基因表现出强大的调控作用。
Para_06
- 总之,我们的研究编制了一个全面的数据集,涵盖了单核水平的基因表达和染色质可及性特征,并伴有五个出生后发育关键时间点的染色质接触图谱。
- 这些数据为调控出生后细胞类型特异性的机制以及其在脑部疾病病因中的潜在作用提供了宝贵的见解。
Methods
Ethical approval
伦理批准
Para_01
- 所有脑组织样本均通过知情同意或通过美国国立卫生研究院(NIH)神经生物银行(马里兰和西奈山脑库)的脑捐赠计划获得。
- 所有程序和研究方案均已获得相关伦理委员会的批准;对于来自西奈山脑生物库的尸检样本,人体受试者保护项目办公室/西奈山医学院机构审查委员会提供了研究程序的指导原则。
- 对于来自马里兰脑生物库的样本,机构审查委员会提供了研究程序的指导原则。
Postmortem brain sample collection details
死后脑样本采集细节
Para_01
- 通过 NIH 神经生物银行(马里兰和西奈山大脑银行),分别获取了人类出生后的脑组织样本。
- 所有神经心理学、诊断和尸检协议均经过各自机构审查委员会的批准。
- 样本来自总共十名对照捐赠者的四个脑区(ACC、DLPFC、HIPP、CN),每个年龄段组别各有一名男性和女性捐赠者(年龄组(岁):婴儿期(0 岁)、中童年期(4 岁和 6 岁)、青春期(14 岁)、青年期(20 岁和 39 岁)以及老年期(60 岁和 61 岁))。
FANS sorting of nuclei for multiome single-nuclear assays
用于多组学单核检测的细胞核荧光激活分离技术
Para_01
- 样本按照性别和年龄随机化,25毫克冷冻的人类死后脑组织在冷裂解缓冲液(0.32 M 蔗糖,5 mM 氯化钙,3 mM 醋酸镁,0.1 mM EDTA,10 mM Tris-HCl (pH 8),1 mM 二硫苏糖醇(DTT)和 0.1% Triton X-100)中均质化,随后通过40微米滤网过滤。
- 滤液覆盖在蔗糖溶液(1.8 M 蔗糖,3 mM 醋酸镁,1 mM DTT 和 10 mM Tris-HCl (pH 8))上,并在4°C下以107,000g离心1小时。
- 沉淀用含0.5%牛血清白蛋白(BSA)的PBS重悬。
- 在进行荧光激活核分选(FANS)之前,用PBS将体积调整至250微升,并根据制造商说明加入7AAD(Invitrogen);7AAD阳性的核被分选到预先涂有5% BSA的管中,使用FACSAria流式细胞仪(BD Biosciences)进行分选(分选策略请参见扩展数据图1b–d)。
Multiome ATAC-seq and gene expression library preparation
多组学 ATAC-seq 和基因表达文库构建
Para_01
- 按照 FANS 方法,细胞核用 200 微升洗涤缓冲液(10x Genomics)洗涤两次,然后重新悬浮于 30 微升细胞核缓冲液(10x Genomics)中,并进行定量(Countess II,Life Technologies)。
- 每份样本中的 8,000 个细胞核使用 Chromium Next GEM 单细胞多组学 ATAC 和基因表达协议(10x Genomics)进行处理。
- 所得文库使用 KAPA 文库定量试剂盒(KAPA Biosystems)进行定量,并通过 Tapestation(Agilent)测定片段大小。
- 所有文库都在纽约基因组中心使用 Illumina Novaseq 600 平台进行测序,其中 snRNA-seq 测序采用配对末端配置,读长分别为 28 碱基对(bp)的第 1 读段和 90 bp 的第 2 读段。
- 对于 snATAC-seq,测序采用配对末端配置,读长分别为 50 bp 的第 1 读段、16 bp 的第 2 读段(包含条形码和唯一分子标识符(UMI))以及 49 bp 的第 3 读段。
FANS sorting of nuclei for Hi-C
用于Hi-C的细胞核荧光激活核分选(FANS)
Para_01
- GABA能神经元(DAPI+NeuN+SOX6+)、谷氨酸能神经元(DAPI+NeuN+SOX6−)、少突胶质细胞(DAPI+NeuN−SOX10+)和小胶质细胞/星形胶质细胞(DAPI+NeuN−SOX10−)的细胞核被分选到单独的试管中(预先涂有5% BSA),分选使用FACSAria流式细胞仪完成。
- 分选策略请参见扩展数据图1b–d。
Hi-C library preparation
Hi-C文库制备
Para_01
- 在FANS之后,大约从每个亚群中提取了10万个细胞核,包括GABA能神经元(DAPI+NeuN+SOX6+)、谷氨酸能神经元(DAPI+NeuN+SOX6−)、少突胶质细胞(DAPI+NeuN−SOX10+)和小胶质细胞/星形胶质细胞(DAPI+NeuN−SOX10−),并按照制造商的说明使用Arima-HiC试剂盒哺乳动物细胞系指南(货号A51008)生成Hi-C文库。
- 根据制造商的说明,使用贝克曼库尔特AMPure SPRIselect磁珠纯化样品。
- 样品立即使用Covaris S220进行超声处理,目标片段大小为300-500 bp,然后根据制造商的说明通过贝克曼库尔特AMPure SPRIselect磁珠进行尺寸选择和纯化。
- 随后,根据制造商的说明,使用Arima-HiC试剂盒和Swift Biosciences Accel-NGS® 2S Plus DNA文库试剂盒对样品进行生物素富集以制备文库。
- 根据制造商的说明,使用Swift Biosciences Accel-NGS 2S Plus DNA文库试剂盒(货号21024)进行末端修复和接头连接。
- 根据制造商的说明,从Swift Biosciences 2S Indexing Kit(货号26148)中为每个样品连接唯一的索引。
- 使用Kapa Hyper Prep Kit(货号NC0709851)和贝克曼库尔特AMPure SPRIselect磁珠,根据制造商的说明对DNA文库进行扩增和纯化。
RNAscope in situ hybridization
RNA原位杂交
Para_01
- 使用 RNAscope 原位杂交技术对人类齿状回的 20 微米厚冰冻切片进行了检测(来自一位 35 岁男性对照捐赠者的两个样本),以验证转录上不同的 OPC 和 OL 细胞群。
- 组织切片通过 Microm HM 505 冷冻切片机制备,并安装在 SuperFrost Plus 显微镜载玻片(Thermo Fisher Scientific)上。
- RNAscope 探针由 Advanced Cell Diagnostics (ACD) 设计和合成,用于靶向感兴趣的 mRNA,包括 PDGFRA 和 ATRNL1(分别标记 I 型和 II 型 OPC)、BMPER(标记 II 型 OPC)、GPR37(标记 I 型 OL)、RBFOX1、以及 ACTN2(标记 II 型 OL)(扩展数据图 6)。
Para_02
- 根据制造商的说明,使用 RNAscope Multiplex Fluorescent Reagent Kit v.2(ACD)进行杂交,并稍作修改。
- 简而言之,切片用 PBS 中的 4% 多聚甲醛(Electron Microscopy Sciences)固定,并用梯度乙醇溶液脱水。
- 然后在切片周围创建疏水屏障。
- 为了降低背景信号,将过氧化氢应用于切片。
- 之后,切片用 Protease IV 处理以消化组织,从而促进探针的渗透。
- 预处理完成后,切片与基因特异性探针混合物孵育,随后与扩增试剂(AMPs)孵育,并通过与辣根过氧化物酶探针和稀释的荧光团孵育来发展荧光信号。
- 杂交后,切片用 4′,6-二脒基-2-苯基吲哚(DAPI;Thermo Fisher Scientific)复染以可视化细胞核。
- 使用 EVOS M7000 显微镜,在 ×20 放大倍率下(Olympus ×20 物镜,氟化物,数值孔径 0.45/工作距离 6.6–7.8)捕获齿状回的图像。
- 使用 ImageJ(v.1.54j)对图像进行拼接和分析,以实现目标 mRNA 的共定位。
Quantification of multiome ATAC-seq and gene expression
多组学ATAC-seq和基因表达的定量分析
Para_01
- 使用 cellranger-arc(版本 1.0.0)进行了 Fastq 文件的比对、过滤、条形码计数、峰呼叫以及 RNA 和 ATAC 片段的计数。
Quality control and data processing
质量控制与数据处理
Para_01
- 最初,使用 King 软件(v.2.3.2)在每个样本的基础上验证了测序数据,以推断关系并防止元数据不匹配。
- Cell Ranger ARC 的输出结果通过 Seurat(v.5.0.1)和 Signac(v.1.12.0)进行处理,生成包含每个样本基因表达和染色质可及性特征的多组学 Seurat 对象。
- MACS2(v.2.2.7.1)作为 Signac 中 CallPeaks 函数的一部分,用于从对应的片段文件中调用峰。
- 所有位于非标准染色体上或与 hg38 基因组注释的黑名单区域重叠的峰均被移除。
- 使用 Signac 中的 FeatureMatrix 函数对每个细胞的每个峰的片段计数进行量化,并随后计算每种模态的单细胞质量控制指标。
- 这些指标包括线粒体比例、核小体信号和转录起始位点(TSS)富集分数。
- 接下来,使用 Seurat 的合并函数将所有单一 Seurat 对象组合成一个包含所有脑区的多模态 Seurat 对象。
- 然后根据两种模态的质量控制指标对细胞进行过滤,保留满足以下条件的细胞:RNA 测序计数 200 < RNA-seq 计数 < 50,000,总 ATAC-seq 计数 200 < ATAC-seq 计数 < 100,000,线粒体比例 < 5%,核小体信号 < 3 和 TSS 富集分数 > 1。
- 此外,检测到少于十个细胞的基因或峰被排除在外,同时使用 Scrublet 标记为双细胞的细胞也被排除,其识别出的细胞占总群体的比例不到 1%。
- 经过对典型细胞类型特异性标志物的仔细检查,我们排除了任何表现出来自不同细胞类型的典型标志物强表达的细胞核,因为它们可能代表中间状态或其他双细胞。
- 最终,保留了 182,818 个单核中的 101,924 个,平均每个细胞有 6,753 个 RNA UMI 计数和 21,876 个独特的 ATAC-seq 片段(33.2% 的片段与峰重叠)。
Para_02
- 使用 SCTransform 方法对基因表达计数进行了归一化,然后使用 Seurat 中的 RunPCA 函数进行主成分分析(PCA)。
- 根据肘部图确定的前 15 个主成分(PCs)被用于统一流形逼近和投影(UMAP)计算。
Para_03
- 使用 Signac 中的 RunTFIDF 函数(参数设置:‘method = 3’),通过词频-逆文档频率 (TF-IDF) 转换的对数-TF 版本对 snATAC-seq 峰值进行归一化。
- 通过 Signac 中的 FindTopFeatures 函数选择 TF-IDF 矩阵中观察频率最高的前 25% 的峰值,并通过 RunSVD 函数进行奇异值分解。
- 在染色质可及性数据中,使用潜在语义索引 (LSI) 进行降维,选取前十个 LSI 成分(排除第一个成分,因为它通常反映技术变异,主要捕获测序深度)用于下游分析。
- 然后通过 Signac 中的 GeneActivity 函数根据 snATAC-seq 数据推断基因活性矩阵,该函数通过评估基因主体和启动子区域的染色质可及性来实现。
Clustering and visualization
聚类与可视化
Para_01
- 在降维后的基因表达和染色质可及性数据上进行了基于图的聚类。
- Seurat(v.5.0.1)中的 FindMultiModalNeighbors 函数被用于多模态分析,通过结合两种模态计算出的降维结果构建了一个加权最近邻(WNN)图。
Para_02
- 为了为单一检测生成共享最近邻图,应用了FindNeighbor函数。
- 最后,在构建的图上使用FindClusters函数(‘algorithm = 3’,‘resolution = 0.2’)利用了Smart Local Moving算法,这在数据集中生成了19个聚类,这些聚类被合并为十种主要的细胞类型(少突胶质细胞、少突胶质前体细胞、星形胶质细胞、小胶质细胞、GABA和谷氨酸能神经元、内皮细胞、VLMC、周细胞和室管膜细胞)。
Para_03
- Seurat 中的 RunUMAP 函数分别应用于基因表达和染色质可及性的主成分和 LSI 组件,以及多组学 WNN 图谱用于联合分析(nn.name = ‘weighted.nn’)。
- RunUMAP 函数实现了 UMAP,可以对多维数据进行二维可视化。
Para_04
- 为了识别每种分类细胞类型的标志物(基因或峰),应用了函数 FindAllMarkers(only.pos = ‘TRUE’,测试 = ROC 和 MAST)。
- 为了提取差异表达基因,进一步添加了参数(min.pct = 0.2,min.diff.pct = 0.1),这些参数确保标志物在相应的细胞簇中显著表达。
- 同样地,对于差异可及的染色质,使用了逻辑回归模型,并将片段总数作为潜在变量加入,这遵循了 Signac 教程系列中的建议(min.pct = 0.05,test.use = ‘LR’,latent.vars = ‘atac_peak_region_fragments’)。
- 最后,调整后的 P 值(Bonferroni 校正)< 0.05 的基因或峰被视为特定细胞类型的基因或峰标志物。
Cluster annotation
聚类注释
Cell-type annotation
细胞类型注释
Para_01
- 使用 Seurat 中的 FindAllMarkers 函数检测到的基因标记以及 SingleR(v.2.2.0)中的自动注释对细胞簇进行了注释(参考数据集为 Allen Brain Atlas 的 M1 皮层 https://portal.brain-map.org/atlases-and-data/rnaseq/human-m1-10x;Cortex/Hipp/Dentate Gyrus 和 Thalamus 参考数据来自 CELLxGENE https://cellxgene.cziscience.com/collections/283d65eb-dd53-496d-adb7-7570c7caa443),并在 Pegasus(v.1.7.1)中使用内置注释(参考数据集为 HB)。
- 通过在簇水平伪批量基因表达谱上计算余弦相似度,验证了标签之间的相关性。
- 细胞类型注释通过多个已发表的数据集进行了验证(扩展数据图 2b,c)。
Subtype annotation in OPC and OL subpopulations
OPC和OL亚群中的亚型注释
Para_01
- 基于 Seurat 中 FindAllMarkers 函数检测到的独特且最富集的标记,对 OPC 和 OL 亚群进行了亚型注释。
Variance partition and cell-type composition
方差分解与细胞类型组成
Para_01
- 方差分解(v.1.31.16)在基因表达的原始计数以及 snATAC-seq 的峰值上运行,以确定数据中变异的驱动因素。
- 公式(1)用于建模:
Para_02
- 将年龄作为固定效应,而个体、性别、脑区域和脑库则被视为随机效应。
- 使用 crumblr 包测试细胞类型特异性的组成变化时,也采用了相同的公式。
Pseudotime analysis on OPC and OL populations
对 OPC 和 OL 群体的伪时间分析
Reconstruction of the oligodendrogenesis trajectory with monocle3
使用 Monocle3 重建少突胶质细胞生成轨迹
Para_01
- 为了概括覆盖多种 OPC 和 OL 群体的少突胶质细胞生成的多组学谱,使用联合 UMAP 整合 RNA 和 ATAC 模态上的细胞-细胞相似性,并通过 monocle3(v.1.3.1)构建主图。
- 具体而言,使用 SeuratWrappers(v.0.3.5)将 Seurat 对象转换为 cell_data_set 对象,然后运行 monocle3 中的函数,包括 preprocess_cds、cluster_cells 和 learn_graph。
Para_02
- 为了将主图投影回单细胞数据,我们为每个细胞识别了最近的顶点,并计算了每个主图顶点的聚类百分比。
- 选择具有最高 OPC I 型百分比的顶点作为伪时间的根节点。
- 然后在 cell_data_set 对象上调用 order_cells 函数,每个细胞获得一个伪时间值,表示其沿少突胶质发生过程的发育进展。
- 此伪时间排序随后用于计算与 SCENIC+ 中的 eRegulon 丰富度的皮尔逊相关性。
Reconstruction of the oligodendrogenesis trajectory with Palantir
使用 Palantir 重建少突胶质细胞生成轨迹
Para_01
- Palantir(版本1.3.3)被应用于OPC/OL群体,使用1200个随机选择的航点,并选择一个OPC细胞(年龄最小且PDGFRA表达最高的细胞)作为起点。
- 参数k(用于构建最近邻图的邻居数量)设置为数据集中细胞总数的10%。
- 扩散成分的数量根据扩散算子特征向量分解的特征间隙确定,这遵循标准的Palantir教程。
Reconstruction of the oligodendrogenesis trajectory with PC1
使用主成分1(PC1)重建少突胶质细胞生成轨迹
Para_01
- 主成分分析(PCA)被用于通过根据细胞年龄对其标记来计算伪时间,从而让我们能够可视化主成分在分化轨迹上分离细胞的效果。
Geneset enrichment analysis
基因集富集分析
Para_01
- 使用 enrichR(v.3.2)和 clusterProfiler(v.4.14.0)包以默认参数运行了基因集富集分析(GSEA)。
- 基因集富集是通过以下参考基因集数据库计算的:GO_Molecular_Function_2021、GO_Biological_Process_2021、WikiPathway_2021_Human、Elsevier_Pathway_Collection 和 Reactome_2022。
Hotspot module analysis
热点模块分析
Identification of oligodendrogenesis gene expression modules
少突胶质细胞生成相关基因表达模块的鉴定
Para_01
- 为了提名与少突胶质细胞生成谱系表达模式一致的基因,我们在单细胞 cell_data_set 对象上运行了 monocle3 的 graph_test 函数。
- 我们选择了 Q 值 < 0.01 且 Moran’s I > 0.5 的基因。
- 为了去除检测率低的基因,我们进一步筛选了在单细胞数据中平均表达量 > 0.2 且标准化方差 > 0.8 的基因,以及在 RNA 测序生成的伪批量样本中被检测到的比例超过 5% 的基因。
- 这最终返回了总共 2,600 个候选基因。
- 使用这 2,600 个提名基因的伪批量基因表达谱(总转录本计数)创建了一个 AnnData 对象,并通过 scanpy (v.1.9.4) 进行处理。
- 归一化的 AnnData 对象被用作 Hotspot (v.0.9.0) 的输入,其中 latent_obsm_key = ‘X_pca’ 且 model = ‘normal’。
- 在创建 KNN 图(n_neighbors = 30)、计算自相关和局部相关后,使用 create_module 函数(min_gene_threshold = 50 和 fdr_threshold = 0.05)在 Hotspot 对象上调用了基因模块。
- 最终输出了总计 12 个模块。
Enhancer-driven gene regulatory networks
增强子驱动的基因调控网络
Para_01
- 我们的包含101,924个核的多组学数据集被用于SCENIC+(v.1.0.0)分析。
- 然而,该数据集的规模超出了SCENIC+的标准容量,因此我们对其进行了修改以适应数据集的需求。
- 更具体地说,在pyCistopic中,我们将区域划分为每个染色体,然后使用‘find_highly_variable_features’和‘find_diff_features’函数识别细胞类型之间的差异可及性区域(DARs)。
- 为了在SCENIC+中推断增强子调控网络(eGRN),我们通过‘calculate_regions_to_genes_relationships’函数将区域按染色体划分以推断区域与基因的关系,并通过‘calculate_TFs_to_genes_relationships’函数将基因划分为小集合以推断转录因子与基因的关系。
Para_02
- 我们使用了 pycisTopic(版本 1.0.2)(https://github.com/aertslab/pycisTopic)Python 模块,来计算条形码级别的质量控制统计信息,并从 ATAC 片段文件中过滤条形码。
- 为了质量控制,我们考虑了每个细胞核条形码满足 Log_unique_nr_frag > 3、TSS 富集度 > 2 和 FRIP(位于峰区域的读取比例)> 0.2 的条形码。
- 我们使用细胞类型标签生成每种细胞类型和脑区的伪批量分布图谱,并使用 MACS2(版本 2.2.7.1)调用峰,参数为:–shift 73–ext_size 146–q_value 1 × 10−10。
- 在调用峰之后,通过使用 pycisTopic 中的函数 get_consensus_peaks 并移除黑名单区域,得到了 861,214 个共识峰。
- 接下来,通过使用函数 run_cgs_models(n_iter = 500, alpha = 50, eta = 0.1) 进行主题建模。
- 通过应用函数 evaluate_models 评估理想的主题数量,并优化为 16 个主题。
- 使用细胞-主题概率通过 UMAP 进行降维以可视化主题结构(补充图 2b)。
- 通过 otsu 方法对区域-主题概率进行二值化处理,并选取每个主题的前 3,000 个区域。
- 为了识别差异可及区域(DARs),我们使用函数 impute_accessibility 以 scale_factor = 10−6 对区域可及性矩阵进行插值,并对可及性矩阵进行 log-标准化。
- 接下来,通过使用函数 find_diff_features 对每种细胞类型和脑区进行分析,基于 Wilcoxon 秩和检验对插值概率矩阵进行测试,并选择 logFC > 0.58 且 Benjamini–Hochberg 校正 P 值 < 0.05 的区域作为 DARs。
Para_03
- 随后,通过利用 pyCistarget(v.1.0.2)Python 模块进行了基序富集分析。
- 我们使用了预先计算的数据库,其中包括基因组区域的基序得分和排名,以及人类基因组(hg38)的基序到转录因子注释数据库。
- 接下来,通过使用 run_pycistarget 函数对每个主题的二值化主题和细胞类型及脑区之间的差异可及性区域(DARs)进行了基序富集分析。
Para_04
- 在遵循 pyCistopic(v.1.0.0)和 pyCistarget(v.1.0.2)的基础上,使用了 SCENIC+(v.1.0.0)(https://github.com/aertslab/scenicplus)Python 模块,通过结合基因表达和染色质可及性数据推断增强子调控网络(eGRNs)。
- 首先,创建了一个 SCENIC+ 对象以进行下游分析,并通过 merge_cistromes 函数生成顺式作用元件集合,该函数将从基序富集字典中分配给转录因子的目标基因与 SCENIC+ 对象中的峰区域重叠。
- 接着,使用 calculate_regions_to_genes_relationships 函数结合梯度提升机计算增强子到基因对模型。
- 然后,基于基因表达数据,使用 calculate_TFs_to_genes_relationships 函数结合梯度提升机推断转录因子与目标基因之间的关系。
- 最后,通过 SCENIC+ 的 build_grn 函数采用恢复方法构建 eGRNs,参数设置为:min_target_genes = 10, adj_pval_thr = 1, min_regions_per_gene = 0, quantiles = (0.85, 0.90, 0.95), top_n_regionTogenes_per_gene = (5, 10, 15)。
- 对于每个增强子调控模块(eRegulon),使用 AUCell 算法计算目标基因和区域的细胞富集分数(AUC)。
- 进一步应用过滤标准,包括选择激活型 eRegulon 和设定基因及区域 AUC 的皮尔逊相关系数阈值 >0.32。
- 此过程共鉴定出 146 个 eRegulons。
Para_05
- 为了确定细胞类型特异性的eRegulons,我们使用函数regulon_specificity_scores计算了细胞类型之间的RSS,并展示了每种细胞类型的前三个到五个调控子(图2b)。
- 为了识别与细胞类型和年龄状态相关的特定eRegulons,类似的方法被应用于选定发育阶段的细胞类型特异性eRegulons(补充图3)。
Para_06
- 使用 TOBIAS (v.1.10.0) 进行足迹分析,以评估开放染色质区域中转录因子的结合活性。
- snATAC-seq 数据的 BAM 文件通过 TOBIAS 的 ‘ATACorrect’ 功能进行处理,以校正 Tn5 转座酶偏差,随后使用 ‘ScoreBigwig’ 和 ‘FootprintScores’ 功能计算每个转录因子的足迹分数。
- 然后对不同样本和条件下的转录因子活性差异进行量化和可视化,从而识别参与细胞类型特异性基因调控的关键调控元件。
Overlap with common genetic risk variants
与常见遗传风险变异重叠
Para_01
- 我们评估了细胞特异性(标志物)峰和基因在十种与大脑相关的特征中的潜在参与,这些特征代表了最常见的神经退行性疾病和神经精神疾病。
- 为了找到每种已识别细胞类型的标志物(基因或峰),我们使用了 FindAllMarkers 函数(only.pos = TRUE)。
- 对于基因表达标志物,我们进一步使用了参数(min.pct = 0.2, min.diff.pct = 0.1),以确保基因在相应的细胞簇中充分表达。
- 对于染色质可及性标志物,我们使用了逻辑回归模型,并将片段总数作为潜变量添加,这在 Signac 教程中有所建议(通过指定进一步的参数:min.pct = 0.02, test.use = ‘LR’, latent.vars = ‘atac_peak_region_fragments’)。
- 经过 Bonferroni 校正后调整的 P 值 < 0.05 的基因或峰被保留为细胞类型特异性标志物。
- 由于检测到的标志物峰/基因数量不足,周细胞/VLMC 细胞簇被排除在分析之外。
Para_02
- 对于单细胞核 ATAC 测序 (snATAC-seq) 数据,针对每种细胞类型的标志峰(在两个方向上各增加 500 bp 的填充以包含附近的变异)使用 LD-score 分区遗传力方法进行了分析。
- 该方法计算了细胞类型特异性峰内的常见遗传变异对遗传力的贡献是否比这些区域外的变异更大,并根据变异的数量以及测试峰的遗传背景(例如内含子、外显子、启动子或基因间区域)进行调整。
- 类似地,对于单细胞核 RNA 测序 (snRNA-seq) 数据,使用 MAGMA(v.1.07b)对每种细胞类型的标志基因进行了分析,包括上游 35 kb 和下游 10 kb 的填充以包含近端调控区域中的变异。
- 由于其广泛且复杂的连锁不平衡结构,分析排除了 MHC 区域(hg19 坐标:chr6 上 25–35 Mb)。
- 对图中所有测试应用了假发现率 (FDR)(Benjamini-Hochberg 方法)校正以修正多重检验问题。
- 此分析的结果及汇总统计数据来源提供于补充数据 7 和 16 中。
Para_03
- 除了对疾病中细胞类型富集的一般研究外,我们还应用了MAGMA和S-LDSC方法来探索全基因组关联研究(GWAS)信号在细胞特异性转录因子(TF)调节子中的富集情况。
- 对于这两种方法,我们采用了与之前相同的分析设置。
- 为了防止MAGMA分析中P值的高估,我们将与转录因子调节子相关的目标基因完整集合作为背景集合纳入分析。
Prediction of enhancer–gene interactions
增强子-基因相互作用的预测
Para_01
- 我们使用了基于接触的活动模型(ABC,v.0.2)来重建每个供体的全面调控图谱,该图谱描述了E-P相互作用,利用了供体的染色质可及性数据(在脑区和细胞类型水平)和Hi-C数据(细胞类型水平)(补充图4c)。
- 决定不使用脑区水平的Hi-C数据,而是合并来自相同发育时期的脑区和供体的Hi-C数据(补充图4c),这是由于Hi-C数据在脑区间的变异性较低,并且数据缺失率相对较高,因为我们没有41%(200个中的81个)供体的细胞类型和脑区组合的Hi-C数据。
- 由于小胶质细胞和内皮细胞与主要的神经元和胶质细胞亚群之间存在显著差异(图1b),并且我们的供体级别的Hi-C数据对于这些细胞类型不可用,因此我们决定重新处理现有的小胶质细胞和内皮细胞数据。
Para_02
- 根据作者的指导原则,我们过滤掉了增强子-启动子(E-P)链接:(1) ABC评分<0.015的链接,(2) 与普遍表达基因及Y染色体上的基因相关的链接,以及(3) 在分析的细胞类型中未表达基因的链接。
- 然后,为了缩小对全基因组关联研究(GWAS)性状的关注范围,我们决定只保留与GWAS指数单核苷酸多态性(SNP)或其连锁不平衡伙伴(R2≥0.8)重叠的E-P链接,最终得到41,919个E-P链接。
- 为了限制每个GWAS位点的因果基因数量,我们采用了ABC-MAX方法,允许每个峰只有一个E-P链接,从而得到2,318个E-P链接。
Statistics and reproducibility
统计学与可重复性
Para_01
- 数据收集和分析并非对实验条件盲化。
- 相关时,样本量(n)会在图中或图例中提供。
- 没有使用统计方法预先确定样本量,但在适用的统计测试中考虑了适当的样本量。
- 没有任何数据被排除在分析之外,且实验未进行随机化,核仁的FANS排序除外。
Reporting summary
报告摘要
Data availability
Para_01
- 单细胞核多组学的原始数据可在 NHIM 数据存档中获取,集合编号为 C5371:https://nda.nih.gov/edit_collection.html?id=5371。
- 本文描述的预处理单细胞数据可通过交互式网络浏览器界面 CELLxGENE Discover Census Portal 平台获取,访问链接为:https://cellxgene.cziscience.com/collections/f406a653-c079-4bf9-aab6-85846c27571d,同时也可以在 Synapse 上通过存取号 syn62750396 获取。
- 用于 ABC 计算的 Hi-C 数据已上传至 Synapse,存取号相同,为 syn62750396。
- 小胶质细胞、内皮细胞以及神经元(GABA 能和谷氨酸能)/少突胶质细胞/星形胶质细胞的 Hi-C 数据分别从以下网址下载:https://doi.org/10.7303/syn26207321、https://www.encodeproject.org/experiments/ENCSR507AHE/ 和 https://doi.org/10.7303/syn63888657。
- 突变约束图谱从 https://gnomad.broadinstitute.org/downloads 下载(ExAC: Constraint 部分)。
- Basic Information
- Abstract
- Main
- Results
- Multiome profiling insinuated dominating temporal diversity
- eGRN identifies key postnatal regulators
- Enhancer–gene networks reveal risk loci for brain disorders
- Prioritized enhancer–gene networks identify causal genes in selected brain disorders
- Pseudotime analysis defines key steps in oligodendrogenesis
- Distinct eGRN activation reveals programs in oligodendrogenesis
- Discussion
- Methods
- Ethical approval
- Postmortem brain sample collection details
- FANS sorting of nuclei for multiome single-nuclear assays
- Multiome ATAC-seq and gene expression library preparation
- FANS sorting of nuclei for Hi-C
- Hi-C library preparation
- RNAscope in situ hybridization
- Quantification of multiome ATAC-seq and gene expression
- Quality control and data processing
- Clustering and visualization
- Cluster annotation
- Cell-type annotation
- Subtype annotation in OPC and OL subpopulations
- Variance partition and cell-type composition
- Pseudotime analysis on OPC and OL populations
- Reconstruction of the oligodendrogenesis trajectory with monocle3
- Reconstruction of the oligodendrogenesis trajectory with Palantir
- Reconstruction of the oligodendrogenesis trajectory with PC1
- Geneset enrichment analysis
- Hotspot module analysis
- Identification of oligodendrogenesis gene expression modules
- Enhancer-driven gene regulatory networks
- Overlap with common genetic risk variants
- Prediction of enhancer–gene interactions
- Statistics and reproducibility
- Reporting summary
- Data availability

腾讯云开发者

扫码关注腾讯云开发者
领取腾讯云代金券
Copyright © 2013 - 2025 Tencent Cloud. All Rights Reserved. 腾讯云 版权所有
深圳市腾讯计算机系统有限公司 ICP备案/许可证号:粤B2-20090059 深公网安备号 44030502008569
腾讯云计算(北京)有限责任公司 京ICP证150476号 | 京ICP备11018762号 | 京公网安备号11010802020287
Copyright © 2013 - 2025 Tencent Cloud.
All Rights Reserved. 腾讯云 版权所有